RやPythonは遅いと言われますが、コーディングの初心者にとっては言語自体の特性よりも書いたコードの非効率性の方が問題になることが多いです。今回は系統樹を探索するコードを例に、R言語を用いてどのように効率の良いプログラムが書けるかを試してみました。
今回の目標
今回は系統樹のノードに紐付いた末端のtip数を網羅的にカウントすることを目標とします。例えば以下のような系統樹では
library(treeio)
tree_test <- rtree(10)
plot(tree_test) #Plotting
nodelabels(11:19) #Attatch node labels
ノード14のTip数は2, ノード13のTip数は3, …となります。また、末端のノード=TipのTip数は当然すべて1となります。
いろいろなコードを試してみる
今回テストに用いるデータセットはランダムに生成した系統樹で、Tip数が1000~5000まで1000おきの計5つです。計算にかかった時間とインプットのサイズに対するO(n)をざっくり見積もります。
1. 最も愚直な実装
Rの系統樹解析パッケージtreeioにはoffspringという便利な関数が実装されており、任意のノードに紐付いた子孫のノード群を一括で抽出することができます。そこで最も単純なコードとして以下のようなものが書けます。
ntips_per_node_naive <- function(tree_data){
t <- Sys.time()
#系統樹データをtibble型(tidyverse式のdataframe)に変換
tree_tbl <- as_tibble(tree_data)
#Result vector初期設定
v <- c()
for(n in tree_tbl$node){
#全子孫tipをtibble形式で取得、数をカウントし名前付きvector: v に格納
v[as.character(n)] <- nrow(offspring(tree_tbl,n,self_include=T,tiponly=T))
}
print(Sys.time()-t)
return(v)
}以上のコードの実行時間をテストしてみると
for(L in c(1000,2000,3000,4000,5000)){
set.seed(0)
tree.test <- rtree(L)
print(L)
v <- ntips_per_node_naive(tree.test)
}
#Output
[1] 1000
Time difference of 1.419068 secs
[1] 2000
Time difference of 3.916159 secs
[1] 3000
Time difference of 8.535078 secs
[1] 4000
Time difference of 13.81695 secs
[1] 5000
Time difference of 22.63864 secsこのTip数に対してこの実行時間が長いのかは人によるかと思いますが、入力されたTip数に対して実行時間が線形に推移しないことから、無駄が多いことが想像できます。offspringは便利な関数だけど、引数がtibbleであることから繰り返し呼び出されることは想定されていないのだろうと想像。
2. アルゴリズムを見直す
上の実装では毎回データフレームを読み込んで子孫をすべて呼び出すということをしていたため相当な無駄がありそうです。
系統樹のようなデータ構造では親ノードに紐付いた子ノードがハッシュを用いて即座に呼び出せるため、親から子への対応関係を詰め込んだハッシュを事前に用意しておけばスムーズに探索できます。木構造の探索は効率の良いアルゴリズムが整理されており、ここでは末端にたどり着くまでノードを掘り下げ、末端に着いたらまだ探索していない分岐を掘り下げていく、という深さ優先探索を用いてみました。
ここで深さ優先探索を用いる最も大きなメリットは求めたいデータ(ノードに紐付いたTip数)の性質にあります。
一般に、ノードnに紐付いたTip数T(n)はnの子ノードn+1_a, n+1_bを用いて
T(n) = T(n+1_a) + T(n+1_b)
と表すことができるため、深さ優先探索を用いてルートから順々に木を掘り下げていけば必ずノードがルートからTipにソートされた形で取得でき、その逆順でノードに紐付いたTip数を足し合わせていけば効率よく求めたいデータが取得できそうです。
以上を実装しました。
#vectorから指定の要素を消去する関数
pop_vec <- function(elem,vec){
vec <- vec[!is.element(vec,elem)]
return(vec)
}
#メイン関数
fnc_dfs_Ntips_stack_1 <- function(tree_data,root_node){
starting.time <- Sys.time()
#系統樹データから親子関係のデータフレームを取得
edge_df <- tree_data$edge %>% as_tibble()
#都合によりノード番号を文字型に変更
edge_df$V1 <- as.character(edge_df$V1)
edge_df$V2 <- as.character(edge_df$V2)
#split関数により、親ノードに対する子ノードのvectorを名前付きリストで一括取得できる
parent_to_child_hash <- split(edge_df$V2,edge_df$V1)
#Starting Depth-First Search
#Rootから探索するのでRootをタスクに登録
stack_list <- c(as.character(root_node))
cnt <- 0
#DFS結果を格納するvector
dfs_vector <- c()
while(TRUE){
#残タスクがなくなったら探索終了
if(length(stack_list)==0){break}
#探索するノードをタスクリストから取り出す
node_now <- stack_list[1]
#紐付いた子ノードを取得
child_nodes <- parent_to_child_hash[[node_now]]
#タスクリストから現在のノードを削除
stack_list <- pop_vec(node_now,stack_list)
#探索済みノードをDFS結果に追加
#現在のノードに子供がいれば子ノードをタスクに追加
if(length(child_nodes)!=0){
dfs_vector <- c(dfs_vector,node_now)
stack_list <- c(child_nodes,stack_list)
}else{
dfs_vector <- c(dfs_vector,node_now)
}
}
#探索が終了したら順番を反転させる。こうするとTip→Rootにノードがソートされる
dfs_vector <- rev(dfs_vector)
#Start counting N tips
#結果の名前付きvector
num_tips <- c()
#Tipから順にカウント開始
for(node_now in dfs_vector){
#ノードがハッシュに存在しなければそのノードはTip,つまりTip数=1
#Internal nodeのTip数は子ノードのTip数の和
if(parent_to_child_hash[[node_now]] %>% is.null){
num_tips[node_now] <- 1
}else{
child_nodes <- parent_to_child_hash[[node_now]]
num_tips[node_now] <- num_tips[[child_nodes[1]]] + num_tips[[child_nodes[2]]]
}
}
print(Sys.time()-starting.time)
return(num_tips)
}実行時間をテスト。
for(L in c(1000,2000,3000,4000,5000)){
set.seed(0)
tree.test <- rtree(L)
print(L)
v2 <- fnc_dfs_Ntips_stack_1(tree.test,rootnode(tree.test))
}
#Output
[1] 1000
Time difference of 0.220216 secs
[1] 2000
Time difference of 0.9728699 secs
[1] 3000
Time difference of 1.627984 secs
[1] 4000
Time difference of 2.859491 secs
[1] 5000
Time difference of 4.789272 secs五倍程度速くなったがO(n)は達成できておらず、さらなる改善が見込めます。
3. データの読み込みなど無駄を省く
上のコードは非常に無駄が多く、同じオブジェクトを何度も読んでいました(頭が悪くて最初は上の実装をしてしまった)。個別にテストしようかと思ったけど面倒になってきたので無駄だと思った部分を列挙、全て最適化(?)したバージョンを記載します。
無駄な部分は以下の通り。
- R言語における名前付きリストや名前付きvector(Pythonではdictに対応)は便利だがハッシュ構造が最適化されておらず、fake hashらしい。R言語用のライブラリ
hashを用いてvalueの呼び出しを高速化。 - DFSを全ノードに対して行っていたが、今回はTipに紐付いたTip数は自明に1であるため、Internal node(正確にはRoot+Internal node)のみの探索で十分。
- DFSで探索したノードをvに登録していく際、
v <- c(v,node_now)として追加していたが、この書き方だとオブジェクトvが毎回コピーされて更新されていく。このため探索が進むほどvのサイズが大きくなり非効率的。最初にInternal node数だけの長さを持ったvを準備しておき、インデックスで指定してvの値を埋めていく方が速い。 - タスクリストから探索済みノードを消去する際、pop_vec関数はvector全体を舐めるため遅い。インデックス指定で消去する。
- 返り値のノードに対するTip数も名前付きvectorでなくhash型を使用
以上を踏まえてコードを修正。
library(hash)
fnc_dfs_Ntips_stack <- function(tree_data,root_node){
#系統樹データから親子関係のデータフレームを取得
edge_df <- tree_data$edge %>% as_tibble()
#系統樹データから親子関係のデータフレームを取得
edge_df$V1 <- as.character(edge_df$V1)
edge_df$V2 <- as.character(edge_df$V2)
#split関数により、親ノードに対する子ノードのvectorを名前付きリストで一括取得できる
parent_to_child_hash <- split(edge_df$V2,edge_df$V1)
#名前付きリストをhashオブジェクトに変換
parent_to_child_hash <- hash(parent_to_child_hash)
#Starting Depth-First Search
#Rootから探索するのでRootをタスクに登録
stack_list <- c(as.character(root_node))
#ループのカウントを初期化
cnt <- 0
starting.time <- Sys.time()
#DFS結果を格納するvectorは予めサイズを指定し、indexで指定して値を格納する
dfs_vector <- character(Nnode(tree_data))
while(TRUE){
#残タスクがなくなったら探索終了
if(length(stack_list)==0){break}
#探索するノードをタスクリストから取り出す
node_now <- stack_list[1]
#紐付いた子ノードを取得
child_nodes <- parent_to_child_hash[[node_now]]
#インデックスの1番を毎回取り出しているため、消去もそのインデックスを指定することでオブジェクトの読み込み回数を削減
stack_list <- stack_list[-1]
#Internal nodeに限り探索済みノードをDFS結果に追加、続いてタスクリストに子ノードを追加
if(length(child_nodes)!=0){
cnt <- cnt+1
dfs_vector[cnt] <- node_now #インデックス指定
stack_list <- c(child_nodes,stack_list)
}
}
#探索が終了したら順番を反転させる。こうするとTip→Rootにノードがソートされる
dfs_vector <- rev(dfs_vector)
#Start counting N tips
#tipを取得
tips <- edge_df$V2[! edge_df$V2 %in% edge_df$V1]
#返り値もhash型を使用
num_tips <- hash()
#TipのTip数=1
num_tips[tips] <- 1
starting.time <- Sys.time()
#末端に近いInternal nodeから順にカウント
#Internal nodeのTip数は子ノードのTip数の和
for(node_now in dfs_vector){
child_nodes <- parent_to_child_hash[[node_now]]
num_tips[node_now] <- num_tips[[child_nodes[1]]] + num_tips[[child_nodes[2]]]
}
print(Sys.time()-starting.time)
return(num_tips)
}結果は以下の通り。
for(L in c(1000,2000,3000,4000,5000)){
set.seed(0)
tree.test <- rtree(L)
print(L)
v3 <- fnc_dfs_Ntips_stack(tree.test,rootnode(tree.test))
}
#Output
[1] 1000
Time difference of 0.02190518 secs
[1] 2000
Time difference of 0.06714511 secs
[1] 3000
Time difference of 0.06415701 secs
[1] 4000
Time difference of 0.09432507 secs
[1] 5000
Time difference of 0.131485 secsちゃんとO(n)っぽくなった。
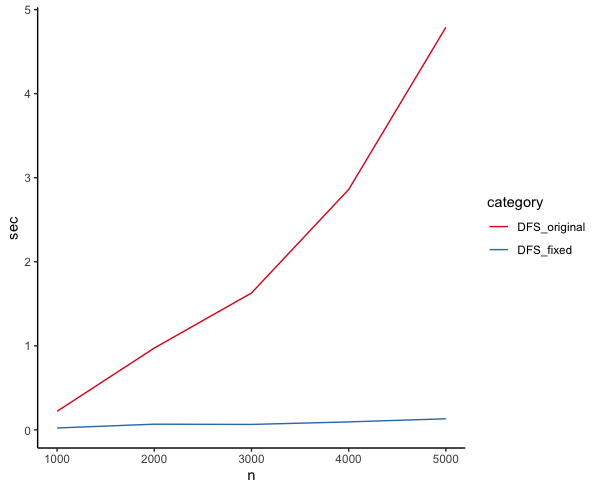
学び
- どの言語にも共通することとして、同じオブジェクトを何度も読み込まない。
- アルゴリズムを正しく理解することで無駄が減る。
- 言語の特性を理解する(Rだと名前付きvector/リストが速くない、vectorへの値の追加がコピーになるなど)
なお、Rのhashに関してはもっと速い書き方があるという話も見ました。時間があればそちらも試してみたいですね。