Illuminaのアセンブラとして、最近使っているのはCLC, SPAdes, platanus, MEGAHIT。velvetやSOAPdenovoはk-merのパラメータを振ってみないといけないけど、これらのツールは複数k-merを使ったり自動で決めてくれたりして、ある程度パラメータを弄らなくても良い結果を出してくれる。アセンブルはメモリ使用量によってアセンブルできるかできないかが分かれる。CPUはあまり重要ではない。メモリ使用量の少ない順はMEGAHIT<CLC<<platanus<<<SPAdesの順。
アセンブル結果について、各コンティグのカバレッジを横軸、コンティグ長を縦軸にとったプロットを作成すると、アセンブル結果の状況がわかりやすいので、私はよくこのカバレッジーコンティグ長グラフを使用します。
まずはCLCの結果
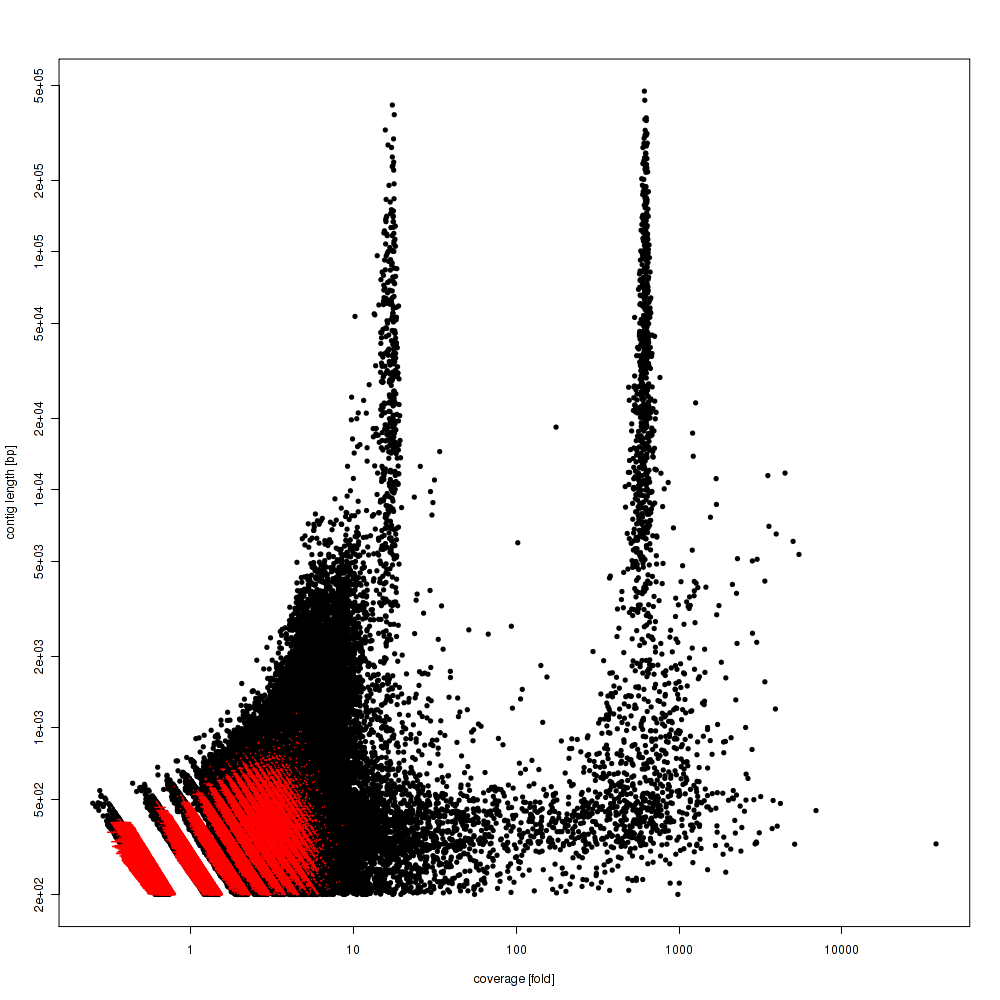
とある寄生虫のゲノムをアセンブルした結果。一番右側の1000x程度のところのピークが目的の寄生虫ゲノム。10x強のところに何かピークが見えるのはおそらくバクテリア。10x弱のところの低いピークは宿主の魚のゲノム。基本的にCLCが一番長く、かつ色々とコンタミしていても綺麗にアセンブルしてくれる印象がある。
次にMEGAHIT
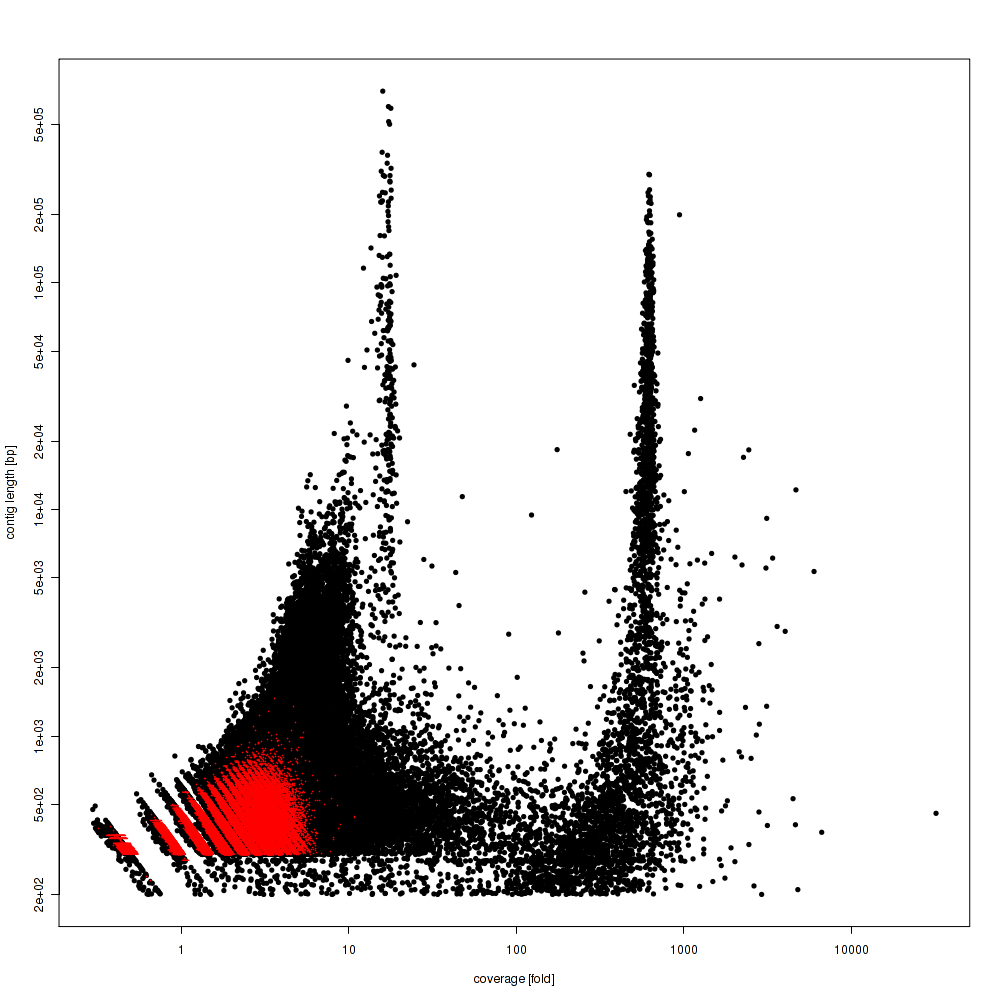
CLCと比べると、目的の寄生虫ゲノムのコンティグ長はだいぶ短い方向にシフトした印象。でも、3つの生物のゲノムがあることはCLC同様にわかる。流石メタゲノム用のアセンブラ。
次にplatanus
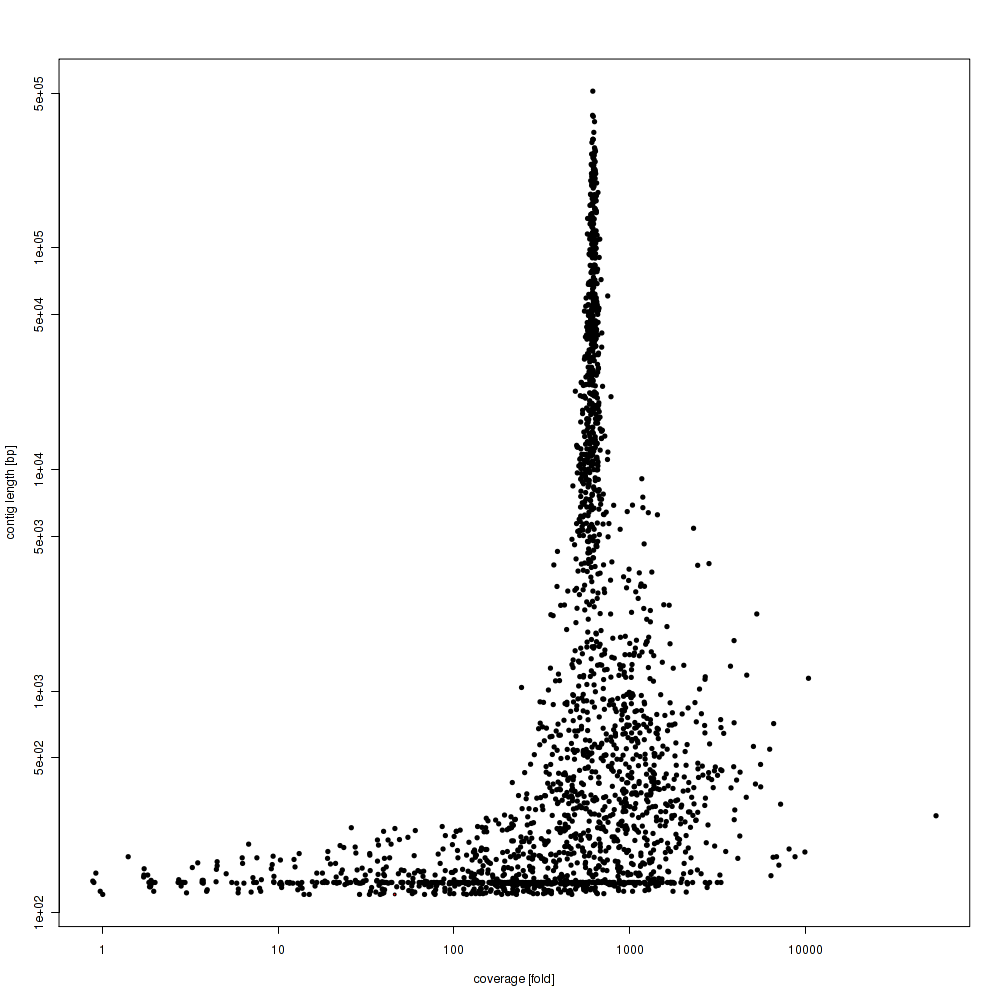
何故か寄生虫のゲノムだけがアセンブルされています。おそらく一番高いk-mer頻度のピークを狙ってアセンブルするように調整されていそうです。このデータの場合は最も寄生虫ゲノムのコピー数が大きかったので問題ないというか、その後寄生虫ゲノムを抽出する手間が省けて良いですが、もし狙っている生物のコピー数がコンタミしている他の生物よりも小さいならば使用できません。寄生虫ゲノムの長さはCLCと同程度でしょうか。
最後にSPAdesの結果。–metaオプションを使った場合、つけない時よりもメモリを使うようで、500 GBでメモリ不足となったので–metaモードは諦めました。
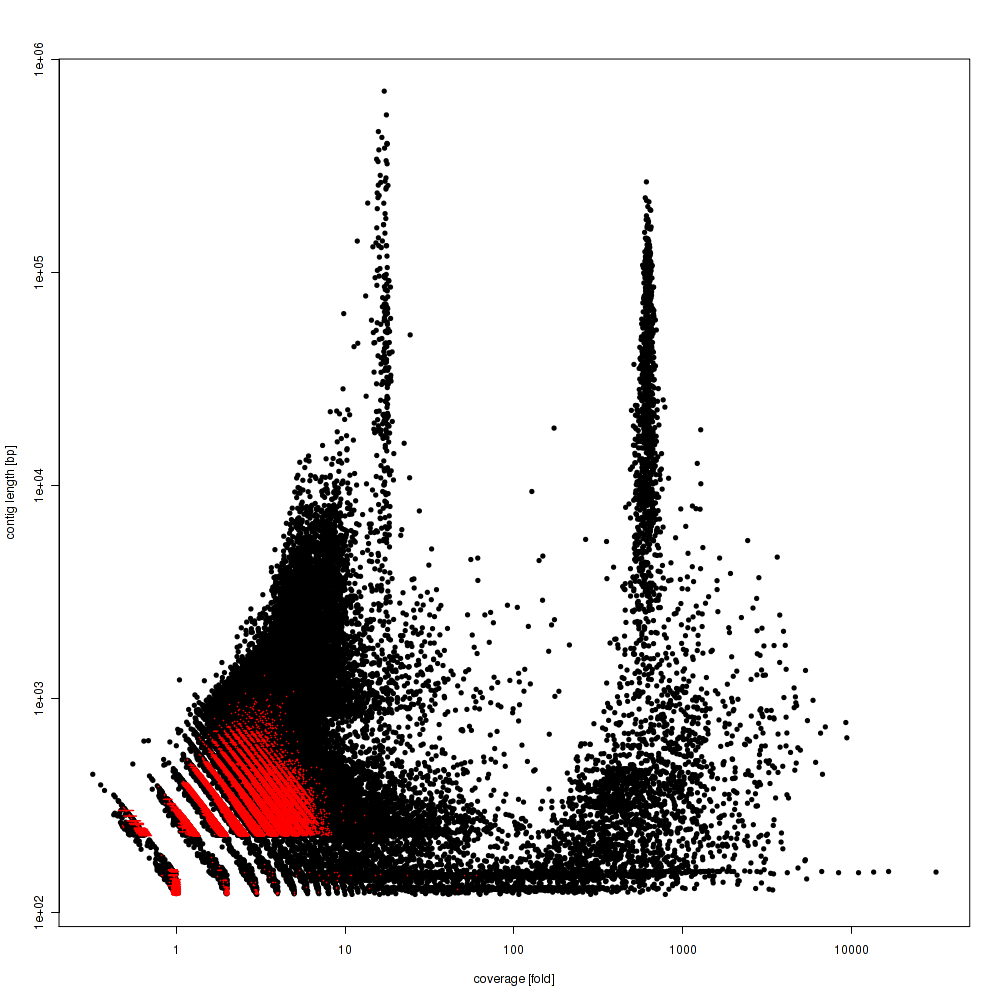
metaモードではないからか、ちょっと宿主ゲノムの分布が変ですが、まぁ許容内でしょう。SPAdesはバクテリアのゲノムアセンブルでよく使われる印象がありますが、10x強のところのバクテリアゲノムは4種の中で最も長く繋いでくれています。寄生虫のゲノムはMEGAHIT以上、CLC, platanus以下といったところでしょうか。ただ、CLC, platanusとの差はパラメータ次第なところはありそうです。メモリが最も多く必要なので、そもそも真核生物向けのアセンブラではないような感じもしますが。
「Illumina用アセンブラ比較」への1件のフィードバック