M2の溝端です。今回は16S rRNAデータベースの検証をしました。
バクテリア16S rRNAデータベースは、メタゲノム解析においてblast検索に広く用いられる。
SILVAやGreengenesをはじめとして、多数のデータベースがWeb上で公開されているが、実用面ではどのデータベースが優れたデータセットになっているかはよくわからない。 今回、8種類のバクテリア16S rRNAデータベースを実際のメタゲノムシーケンス(アンプリコン、ショットガン)に適用することで、その性能を比較検証した。
検証に使用したデータベース
1.SILVA (SILVA_138.1_SSURef_NR99_tax_silva_trunc.fasta)
ドイツにある Leibniz Institute DSMZ-German Collection of Microorganisms and Cell Cultures のSILVA teamによって開発・メンテナンスされている。
最終更新は2020年8月24日。
2.Greengenes (gg_13_5.fasta)
アメリカの Lawrence Berkeley National Laboratoryが運用。
最終更新は2019年5月1日。
3.RDP (current_Bacteria_unaligned.fa)
ミシガン州立大学が提供している。
最終更新は2016年9月30日
4.16S-ITGDB (Hsieh et al., 2022, seq_itgdb_seq.fasta)
SILVA, Greengenes, RDPをマージしてキュレーションしたもの。
2022年の論文なので新しめ。
5.NCBI 16S_ribosomal_RNA (16S_ribosomal_RNA.tar.gz)
NCBIが配布している16S rRNAのデータベース。
最終更新は2023年4月4日。
6.NCBI nt
NCBIが配布しているnucleotideデータベース。16S以外も含まれるので圧倒的にサイズが大きい。
最終更新は2023年4月4日。
7.rrnDB (rrnDB-5.8_16S_rRNA.fasta)
ミシガン大学が配布している16S rRNAデータベース。紛らわしいが、RDPを配布しているミシガン州立大学とは別物らしい。
最終更新は2022年6月23日。
8.GRD
理研が提供しているデータベース。
最終更新日はよくわからないが、なんとなく古そう。
使用したクエリー配列
Ocean Monitoring Databaseに登録されている16Sアンプリコンシーケンスとショットガンシーケンスデータを、それぞれ上から10個ずつ取得してきて各先頭10000行を抽出して使用した。
比較検証
各DB/クエリーの組み合わせに関してblastn検索を行い、トップヒットのbit scoreを比較した。
blastn検索
makeblastdb -dbtype nucl -in db.fasta blastn -db db.fasta -query query.fasta -outfmt 6 -num_threads 16 -max_target_seqs 1000 > query_db.fasta.blastn
トップヒット抽出
cat query_db.fasta.blastn|awk -F"\t" 'BEGIN{list["read"]=1;}{if($1 in list){}else{print;}list[$1]=1;}' > query_db.fasta.blastn.tophit
ビットスコアの順位を集計するpythonスクリプトの設計(summary.py)
#このスクリプトは、複数のblast outfmt6形式ファイル(例えば、同一のクエリーに対して異なるデータベースを用いて計算されたもの)を渡すとビットスコアを集計し、その順位を集計して出力する。
#モジュールの読み込み
import pandas as pd
import numpy as np
import sys
from scipy.stats import rankdata
#引数の取得
args=sys.argv
num_args=len(args)
args_array=[]
ranking=[]
#outfmt6ファイルの読み込み。引数として渡した全てのファイルを参照(args[1]~args[num_args-1])
for i in range(1,num_args):
#リード名とビットスコアのみを抽出。
result=pd.read_csv(args[i],sep="\t",header=None).iloc[:,[0,11]].set_axis(["read",args[i]],axis=1)
#一番最初はdataに抽出結果をそのまま代入
if i==1:
data=result
#2番目以降は抽出結果をdataにマージ
else:
data=pd.merge(data,result,how="outer",on="read")
#引数一覧をstring型arrayで作成。あとで順位表のラベルとして使う。
args_array.append(str(args[i]))
#1~num_args-1までの自然数の数列を作成。あとで順位表のラベルとして使う。
ranking.append(str(i))
#Nan(特定のデータフレームでだけビットスコアが欠落=Nohit)はビットスコア0として埋める
data=data.fillna(0)
#各行に対して順位を出力。(num_args-1)×(num_args-1)のデータフレームを作る。列名は順位、行名はインプットファイル名。
output=pd.DataFrame(np.zeros((int(num_args)-1,int(num_args)-1))).set_axis(args_array,axis=0).set_axis(ranking,axis=1)
#リード数取得
num_ind=len(data)
#各行(各リード)に対して順位を計算。
for i in range(num_ind):
#あるリードに対数ビットスコアの大きさをrankdata()で昇順に取得し、降順に変換で順位を取得。このとき、method=maxをつけないと同じ値があるときに平均順位である小数が出力されてしまうので注意。
rank=len(np.array(data.iloc[i,1:])) - rankdata(np.array(data.iloc[i,1:]),method="max")
#引数k+1番目(=outputのk行目)のファイルの該当順位(rank[k])のカウントを一つ増やす。
for k in range(len(rank)):
output.iat[k,rank[k]]=output.iat[k,rank[k]]+1;
#ラベル全長を表示
pd.set_option('display.max_colwidth', None)
#結果の出力
print(output)
blastnファイルの集計
python summary.py `ls *tophit`
結果
アンプリコン・ショットガンデータについて1例ずつ示す。
アンプリコン
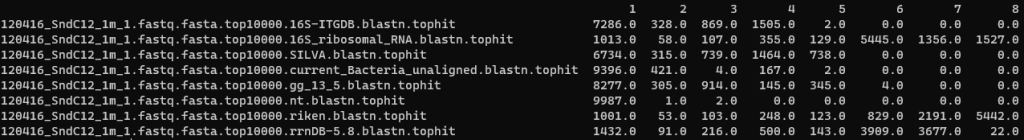
ヒットしたすべてのリードに対して、各DBのbit scoreが1~8位となった回数を集計したもの。同率の場合は同順位としている(例えば同率1位が2つあった場合、上位3つはそれぞれ1位、1位、3位となる)。
最も1位の回数が多いのはnt。9987リードで1位(同率含む)を獲得した。ほかのDBに負けた(=2位以下となった)のはわずか3リードのみであった。
次に優秀なのはRDP。9396リードで1位となった。
Greengenes,16S-ITGDBもそれぞれ8277,7286リードが1位と悪くない。
SILVAは6734リードと予想外に低めだった。
それ以外は1000リード台と、明らかに他と比べて劣る。
ショットガン ←よく見たらショットガンじゃなくてアンプリコンのデータでした
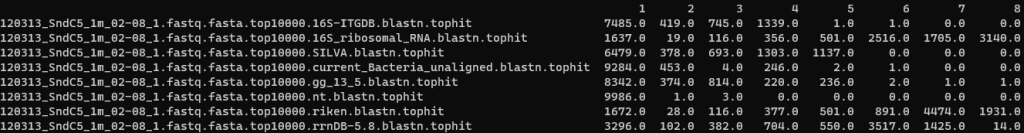
基本的にアンプリコンと変わらない結果となった。ntがぶっちぎり1位でRDPが2位。
そのあとはGreengenes→16S-ITGDB→SILVA→rrnDB→理研→16S(ncbi)の順。
結論(感想)
データベースの性能が最も高い(クエリーに最も近い配列を含む)のはぶっちぎりでNCBIのntデータベースだった。計算資源が無尽蔵にある、あるいはクエリーがそんなに大きくないならntを使うべきだろう。一方、ntは圧倒的にサイズがデカいので、大量のシーケンスデータを処理したい時などには使い勝手が悪い。
16S rRNAのみを含むデータベースとしてはRDPが最も優れており、90%以上のリードでntと同等のパフォーマンスを示した。Taxonomy IDが独特なのが面倒そうだが、性能的にはおすすめである。
よく利用されるSILVAは、(他のDBと比べて)予想外に低パフォーマンスとなった。メタゲノム解析において高精度で種名判定をしたい場合、SILVAはよく検討したうえで利用する必要があるかもしれない。
雑になってしまいましたが、ntやRDPが良くてSILVAがそこまででもない、という結果はなかなか面白かったです。
検証に穴があるかもしれないので、ご指摘・ご意見・感想などございましたら、お気軽にコメントいただけると嬉しいです!
水圏生物工学研究室 M2 溝端
追記
Twitterでこんなご指摘をいただきました。

そこで、SILVAデータベースについて、SILVA_138.1_SSUParc_tax_silva.fastaを使って再検討しました。
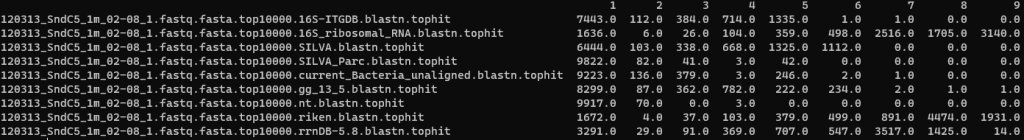
結果、ご指摘の通りSILVAのスコアが飛躍的に改善しました。RDPを超え、ntに迫るスコアとなりました。計算リソースを考えれば、やはりSILVAは優秀なデータベースであると結論できます。計算コストを無視できるのであればntに優位性がありますが、SILVAでも十分な精度がありますし、何よりSILVAの方が取り回しやすいでしょう。データの大きさや計算リソースなど、状況に応じてSILVA,ntを使い分けるべきかもしれません。
windowmoonさん、ご指摘ありがとうございました!