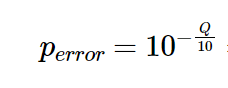**文書の過去の版を表示しています。**
メタゲノム・eDNA実験のデータ解析について
このページのURL https://x.gd/zYXQB
本日は、実際のシーケンスデータを用いた解析実習を行います。コンピュータを使って、メタゲノム解析によって環境中の微生物群集の構造を明らかにしたり、環境DNA解析によって特定の生物種の痕跡を検出したりするバイオインフォマティクス技術を学びます。
メタゲノム解析
環境DNA解析
実験のまとめ(水のサンプリング~DNAシーケンシング)
サンプル
PCR結果
PCR増幅したプライマーの種類
| サンプル | プライマー領域 | 対象生物 | アダプターの付いたPCR断片長 |
| 三四郎池の水 | ミトコンドリア12S rRNA | 魚 | 200 bp強(300 bp強にバクテリア由来の非特異的なバンドも増えることが多い) |
| 三四郎池の水 | バクテリア16S rRNA | バクテリア | 1,500 bp程度 |
| 発酵食品 | バクテリア16S rRNA | バクテリア | 1,500 bp程度 |
1班
2班
3班
4班
シーケンス結果
| ファイル名 | リード数 | 平均リード長 (bp) |
| 1-Bacteria16S-metagenome.fasta | 100000 | 1460.8 |
| 1-Mito12S-eDNA.fasta | 34172 | 168.9 |
| 2-Bacteria16S-food.fasta | 209 | 1414.3 |
| 2-Bacteria16S-metagenome.fasta | 58183 | 1440.2 |
| 2-Mito12S-eDNA.fasta | 99755 | 169.7 |
| 3-Bacteria16S-metagenome.fasta | 73932 | 1442.8 |
| 3-Mito12S-eDNA.fasta | 99822 | 169.3 |
| 4-Bacteria16S-food.fasta | 1216 | 1483 |
| 4-Bacteria16S-metagenome.fasta | 542 | 1448 |
| 4-Mito12S-eDNA.fasta | 99 | 170.4 |
前提知識
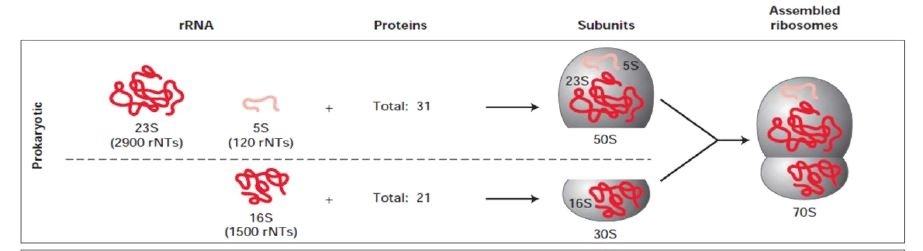
リボソームについて
リボソームはリボソームRNA (rRNA)とタンパク質の複合体であり、原核生物の場合、大きく50Sと30Sのサブユニットに分かれる。30S複合体の中には16S rRNAが含まれる。
16S rRNA
16S rRNAは保存性の高いConserved Regionsと、変化の多いVariable Regionsが交互に出現する。
rRNAの二次構造でVariable Regionsを可視化すると下記のようになる。
各Small Sub Unit (SSU)とLarge Sub Unit (LSU)の名前
| Small Sub Unit (SSU) | Large Sub Unit (LSU) | 補足 | |
| バクテリア | 16S rRNA | 23S rRNA | |
| 真核生物 (核) | 18S rRNA | 28S rRNA | |
| 真核生物 (ミトコンドリア) | 12S rRNA | 16S rRNA | アルファプロテオバクテリア由来 |
| 真核生物 (葉緑体) | 16S rRNA | 23S rRNA | シアノバクテリア由来、バクテリアの16S, 23Sとかなり似ている |
ミトコンドリアの12S rRNAとバクテリアの16S rRNA
ミトコンドリア12S rRNAは真核生物のミトコンドリアにコードされている遺伝子で、バクテリア16S rRNAに相同な遺伝子である。
ミトコンドリア12Sの種間の違い
ミトコンドリア12Sとバクテリア16Sの対応関係
使用したプライマー
- バクテリア16S全長 プライマー部分を除くと1,400-1,500 bp程度のDNA断片
プライマーの名前 配列 バクテリア16S全長 Forward (27F) AGAGTTTGATCMTGGCTCAG バクテリア16S全長 Reverse (1492R) GGTTACCTTGTTACGACTT - ミトコンドリア12S MiFish プライマー部分を除くと170 bp程度のDNA断片
プライマーの名前 配列 ミトコンドリア12S MiFish Forward (MiFish-U-F) GTCGGTAAAACTCGTGCCAGC ミトコンドリア12S MiFish Reverse (MiFish-U-R) CATAGTGGGGTATCTAATCCCAGTTTG 皆さんに配布するシーケンスデータはナノポアのアダプターや上記のプライマー部分をトリミング(削除)してあります。
同種、同属、同科での配列の一致率の閾値
| ミトコンドリア12S MiFish領域 | バクテリア16S全長 | |
| 種 | 98.5-99% | 98.7% |
| 属 | 97% | 94.5% |
| 科 | 95% | 86.5% |
出典:https://journals.plos.org/plosone/article?id=10.1371%2Fjournal.pone.0266720 https://elifesciences.org/articles/85795 https://academic.oup.com/ismecommun/article/1/1/16/7462888
解析の概要
本日のデータ解析のワークフローを以下に示します。
データ解析の大雑把な流れとしては、 各サンプルに含まれていた魚、バクテリアの種類を網羅的に解明します。
- シーケンスデータの品質チェック (AIに作成させたPythonスクリプトで)
- シーケンスデータのクラスタリング&コンセンサス配列作成 (VSEARCH)
- 各リードがどのコンセンサス配列に近いのか調べるために相同性検索 (VSEARCH)、相同性検索結果を集計し、サンプルごとの出現頻度表を作成 (Pythonスクリプト)
- 既存のデータベースを用いて、コンセンサス配列の相同性検索を行い、コンセンサス配列の種名を調べる(VSEARCH)。SILVAは国際的なバクテリア16S rRNAデータベース、MiFishDBは元千葉県立中央博物館の宮博士と佐土博士によって整備されているDB。
- 生物ごとの出現頻度表を作成 (Pythonスクリプト)
配列アラインメント入門
例を用いて、配列アラインメントの考え方と Sum of Pair (SP) スコアの計算までを簡潔に解説します。
配列アラインメントとは?
- 大雑把に言うと 複数の配列を並べたもの。
- 記号 '-' を追加し、似た配列が並ぶようにそろえる。
- 2 本の配列を並べると ペアワイズアラインメント。
- 3 本以上の配列を並べると マルチプルアラインメント。
例) ACATCAGC ||X||| | ACAGCAGC ※ |: マッチ, X: ミスマッチ, -: ギャップ
アラインメントには様々な可能性
例)配列 ACT と配列 ACG を並べる:
ACT A-CT ACT- -ACT ||X | X || |x ACG AC-G AC-G A-CG
見た目では左のものが最も良さそうに見える。計算機に「見た目の良さ」を教えるには、数値的な指標を定義する必要がある。
Sum of Pair (SP) スコア
- 扱いやすいためによく用いられるスコア体系。
- 各列のスコアを 合計 して全体スコアを求める。
| 種類 | スコア |
| マッチ | +3 |
| ミスマッチ | -4 |
| ギャップ | -5 |
例) ACT ||X ACG 列ごとの加点: +3, +3, -4 → 合計 2 点
SP スコアを実際に計算してみる
同じく ACT と ACG を各パターンで並べ、SP スコアを計算:
ACT A-CT ACT- -ACT ||X | X || |x ACG AC-G AC-G A-CG スコア2点 スコア-11点 スコア-4点 スコア-11点 +3 +3 -4 +3 -5 -5 -4 +3 +3 -5 -5 -5 -5 +3 -4
ギャップまで含めると計算すべきアライメントの種類は非常に多いので、取りこぼしがないように網羅的に計算するアルゴリズムが必要。
最も似ているアライメントを効率よく求めるSmith-Watermanアルゴリズム
相同性検索を高速に行うBLASTプログラム
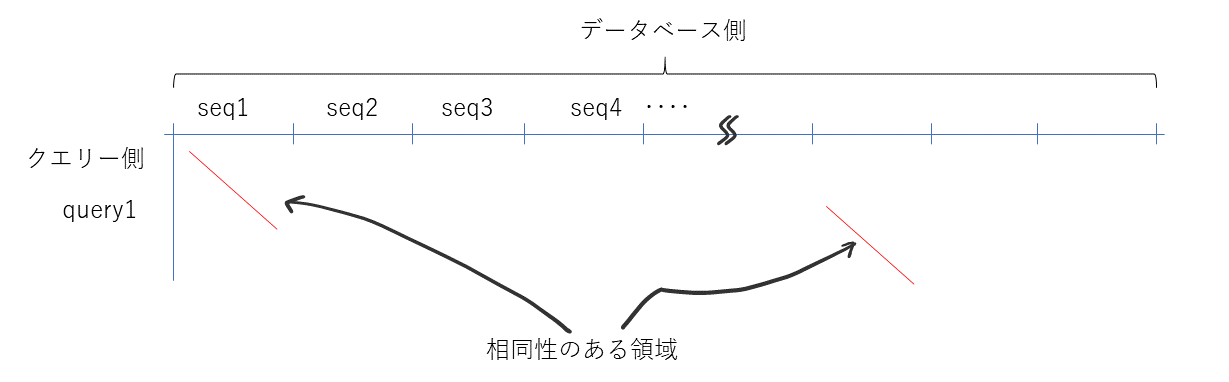
BLASTは、相同性検索(ホモ ロジーサーチ)を比較的高速に行うプログラムである。厳密な解を提供する Smith-Watermanアルゴリズムを少しヒューリスティックにすることで、完全な厳密解は与えないものの実用的には十分な精度を持ちつつ、 Smith-Watermanよりはるかに高速に検索を実現した。 また、BLASTではペアワイズの相同性検索の結果に対して、偶然そのような配列の一致が起こる期待値e-valueを出力し、閾値以上でデータベースとヒットした結果を出力する。
BLASTの高速化の概要
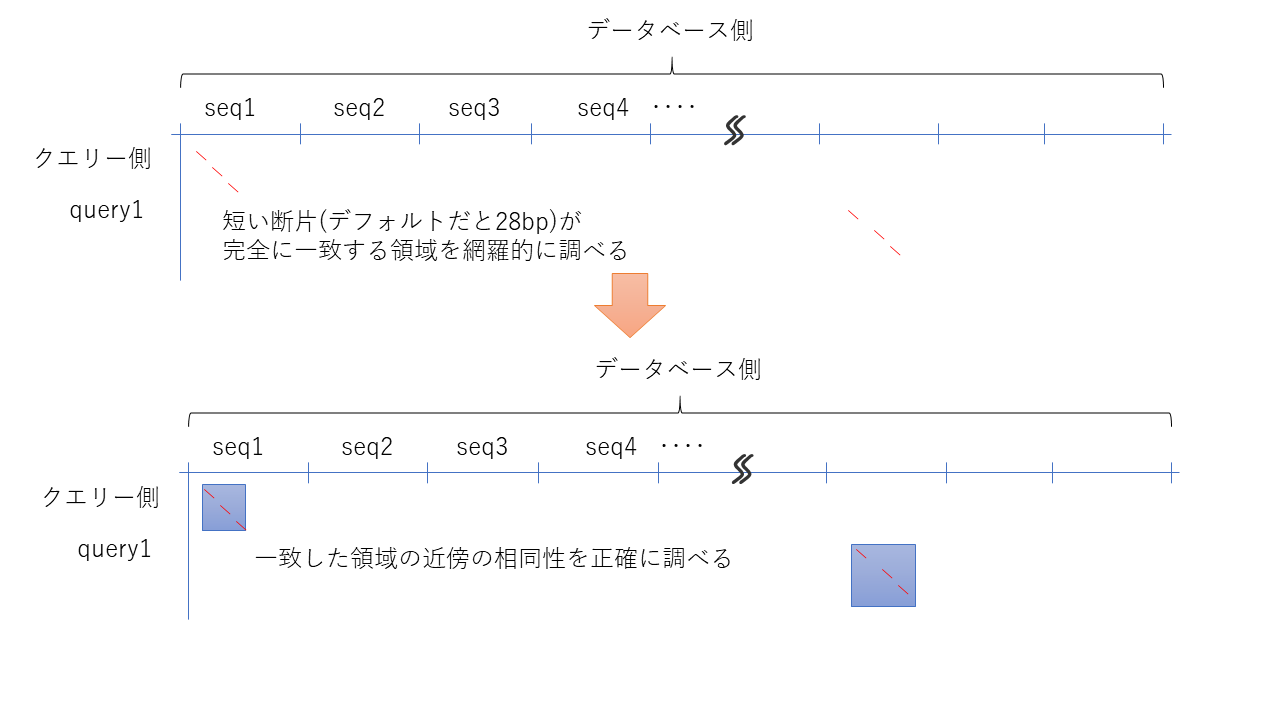
データベース検索する場合、下記のようにデータベース側は100 Gbaseを超える場合もあり、非常に巨大である。しかし、その中で相同な配列というのは通常そんなに多くはない。
そこで、あらかじめwordサイズで指定した大きさで100%マッチする場所を高速に調べておき、その周辺のみ時間をかけて調べることで高速化している。
このwordサイズはNCBIのWEBサイトで公開されているBLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi )などでは、デフォルトが28 bpと比較的大きめなので、シーケンス精度が悪い場合は注意する必要があるかもしれない。
Blastの結果についての説明
https://www.jaici.or.jp/stn/pdf/seqfaq.pdf
BLASTの出力でE-valueは、指数表記で表されるので注意。例えば、「10-4」は「1.0e-4」と表記される。相同性のあるなしに関しての目安としては、(私は)DNAの場合E-valueが1e-10以下、タンパク質の場合1e-5以下であれば相同性がある(かもと思っている)。BLASTではE-valueが1e-150あたりよりも小さくなると「0」として丸めて表示される。 メタゲノム解析ではE-valueよりも一致率のほうが直感的であり、16S rRNAの場合に一般的には98%以上の一致率で同種であると言われる。ただし、分類群によっては属が違っても一致率100%ということがありうる。
BLASTより高速な相同性検索ツール
BLASTよりもさらに高速に相同性検索を行うツールは色々と開発されている。今回使用するVSEARCHはDNAの相同性検索をBLAST以上に高速に実行することが可能である。しかしその反面、BLASTと比べると相同性の低い一致の検出は苦手、基準を満たしたヒットを1件見つけたら終了する、E-valueの計算を行わないといったマイナス要素も存在する。
ツールのインストール
シーケンスデータ、解析スクリプト、既知の配列のダウンロード
data2025←このファイルをダウンロードして解凍しておく。
- Windowsなら
Explorerで「すべて展開」する
- Macなら
ダウンロードしたファイルをダブルクリックすればよい
以下の解析手順は「~/Downloads/data2025」にすべてのデータが展開されていることを前提にしている。異なる場所に展開した場合は適宜変更すること。
解析手順
1. クオリティチェック
一般的に次世代シーケンサー(NGS)のデータは1塩基ごとにクオリティが付与されているが、ナノポアに関しては1塩基ずつのクオリティについては信頼性がなく、リード全体の平均精度として情報が出力されている。
NGSのクオリティとは
クオリティスコアQから、エラーの生じる確率 P(error) は下記のように計算されます。 (出典: https://bi.biopapyrus.jp/rnaseq/qc/fastq-quality-score.html )
- クオリティスコアが 10 ならば、シーケンシングエラーが生じる確率は 10.0% であるから、読み取られた塩基の信頼度は 90.0% 。
- クオリティスコアが 20 ならば、シーケンシングエラーが生じる確率は 1.0% であるから、読み取られた塩基の信頼度は 99.0% 。
- クオリティスコアが 30 ならば、シーケンシングエラーが生じる確率は 0.1% であるから、読み取られた塩基の信頼度は 99.9% 。
| クオリティスコア | シーケンス精度 |
| Q10 | 90% |
| Q13.01 | 95% |
| Q13.98 | 96% |
| Q15.23 | 97% |
| Q16.99 | 98% |
| Q18 | 98.4% |
| Q19 | 98.7% |
| Q20 | 99% |
| Q30 | 99.9% |
シーケンスデータ(FASTA形式)
シーケンスデータのクオリティを確認する方法
ターミナルで下記を実行してダウンロードしたデータのフォルダーに移動する。
cd ~/Downloads/data2025
Geminiを実行する。
gemini
> reads/1-Mito12S-eDNA.fasta はナノポアのシーケンスデータです。リード名の行にqs:f:で始まる項目があり、そのリードのクオリティが書かれています。このシーケンスデータのリード長vsクオリティの図を描くスクリプトを作って。リード長とクオリティのヒストグラムもつけて。不足しているツールは自動でインストールし、作成したスクリプトの使い方を説明して。
下記のようにファイルを保存してよいか、コマンドを実行してよいか尋ねてくるのでYesを押す。Yesを押すのが面倒になったら「Ctrl-y」を押すと承認不要で自動で進めてくれるYOLOモードに変更できる。(ただし、スクリプト作成完了後に不要な提案をして自動で実行しようとすることがあるので、適当なところで止めたほうが良いことも…)
2. クラスタリング
a. まずeDNA、メタゲノムごとにシーケンスデータをひとつのファイルにまとめる。ここではeDNAの場合を説明する。
Windows
cat .\reads\*eDNA.fasta | sc all-eDNA.fasta
Mac
cat ./reads/*eDNA.fasta > all-eDNA.fasta
メタゲノムの場合は、*eDNA.fastaを*metagenome.fastaもしくは*food.fastaに変更し、all-eDNA.fastaもall-metagenome.fasta, all-food.fastaなどと適当に変更すること。
b. まとめたデータをクラスタリングする
--cluster_fast:入力された塩基配列を長さ順にソートしてクラスタリングを行う。クラスターの代表配列としてリード長の長いリードが出力される。
--id:同一クラスターとみなす一致率の閾値
--sizeout:クラスターに含まれるリード数を出力
--consout:クラスターのコンセンサス配列を出力
vsearch --cluster_fast all-eDNA.fasta --id 0.98 --sizeout --consout consensus1.fa
c. シーケンスエラーで1リードだけからなるクラスターが大量に存在するので、下記のコマンドでゴミクラスターを削除
python3 filter_consensus_by_cluster_size.py --min-size 10 consensus1.fa consensus1.min10.fa
d. コンセンサス配列を再度クラスタリングすることで、シーケンスエラーをさらに除去
--cluster_size:クラスターの大きさ順にソートしてからクラスタリング。クラスターサイズの大きい配列が出力される。
--centroids:クラスターの代表配列を保存。
--id:一致率の閾値
vsearch --cluster_size consensus1.min10.fa --centroids consensus2.fa --id 0.98
3. サンプルごとのクラスターサイズ集計
a. コンセンサス配列にシーケンスデータをマッピングし、各リードがどのクラスター由来なのか調べる
--usearch_global, --db:dbで指定したファイルに対して相同性検索を行う。
vsearch --usearch_global reads/1-Mito12S-eDNA.fasta --db consensus2.fa --id 0.9 --blast6out 1-Mito12S-eDNA.fasta.bl6 vsearch --usearch_global reads/2-Mito12S-eDNA.fasta --db consensus2.fa --id 0.9 --blast6out 2-Mito12S-eDNA.fasta.bl6 vsearch --usearch_global reads/3-Mito12S-eDNA.fasta --db consensus2.fa --id 0.9 --blast6out 3-Mito12S-eDNA.fasta.bl6 vsearch --usearch_global reads/4-Mito12S-eDNA.fasta --db consensus2.fa --id 0.9 --blast6out 4-Mito12S-eDNA.fasta.bl6
b. 上で調べた結果を一覧表にまとめる
python3 summarize_top_hits.py 1-Mito12S-eDNA.fasta.bl6 2-Mito12S-eDNA.fasta.bl6 3-Mito12S-eDNA.fasta.bl6 4-Mito12S-eDNA.fasta.bl6 -o abundance.tsv
4. コンセンサス配列の生物種名を調べる
基本的にはvsearchを動かすだけなのだけど、日本語の和名を入れたDBを使用しているためVSEARCHが大量に警告を出してWindowsだと特に遅くなるので下記のように警告を表示しないようにして実行。
Windows
cmd /c "vsearch --usearch_global consensus2.fa --db MiFish_DB_v50_jap.fasta --id 0.9 --blast6out consensus2.fa.bl6 2> warning.txt"
Mac
vsearch --usearch_global consensus2.fa --db MiFish_DB_v50_jap.fasta --id 0.9 --blast6out consensus2.fa.bl6
上記は魚の12S rRNA配列のデータベースなので、メタゲノムの場合は下記の通りSILVAのデータベースを使用する。
Windows
cmd /c "vsearch --usearch_global consensus2.fa --db silva_v138.2.fasta --id 0.9 --blast6out consensus2.fa.bl6 2> warning.txt"
Mac
vsearch --usearch_global consensus2.fa --db silva_v138.2.fasta --id 0.9 --blast6out consensus2.fa.bl6
5. 生物種ごとの出現頻度を集計
クラスターの集計表では、シーケンスエラーによって本当は同一生物由来なのに別クラスターになっているクラスターが存在する。そうした別クラスターを一つにまとめたい。まず、上で得られた生物種名を一覧表に追加する。
python3 add_sequence_column.py abundance.tsv consensus2.fa consensus2.fa.bl6 -o abundance_with_seq_and_blast.tsv
サンプルごとの出現頻度をリード数ではなく%表示に変換し、平均出現頻度の多いクラスターから順番に出力。
python3 normalize_abundance.py abundance_with_seq_and_blast.tsv abundance_normalized.tsv
生物種ごとにカウントをマージ
python3 deduplicate_abundance.py abundance_normalized.tsv
コマンド入力時の便利キー
| カーソルの上・下キー | 前に入力したコマンドを呼び出す。 |
| (Win) Ctrl+Shift+V or 右クリック, (Mac) Cmd+V | 貼り付け |
| tabキー | ファイル名・コマンドの自動補完 |
| Ctrl+C | コマンド強制終了(blastを実行中に止めたい場合など) |
6. Excelでの解析
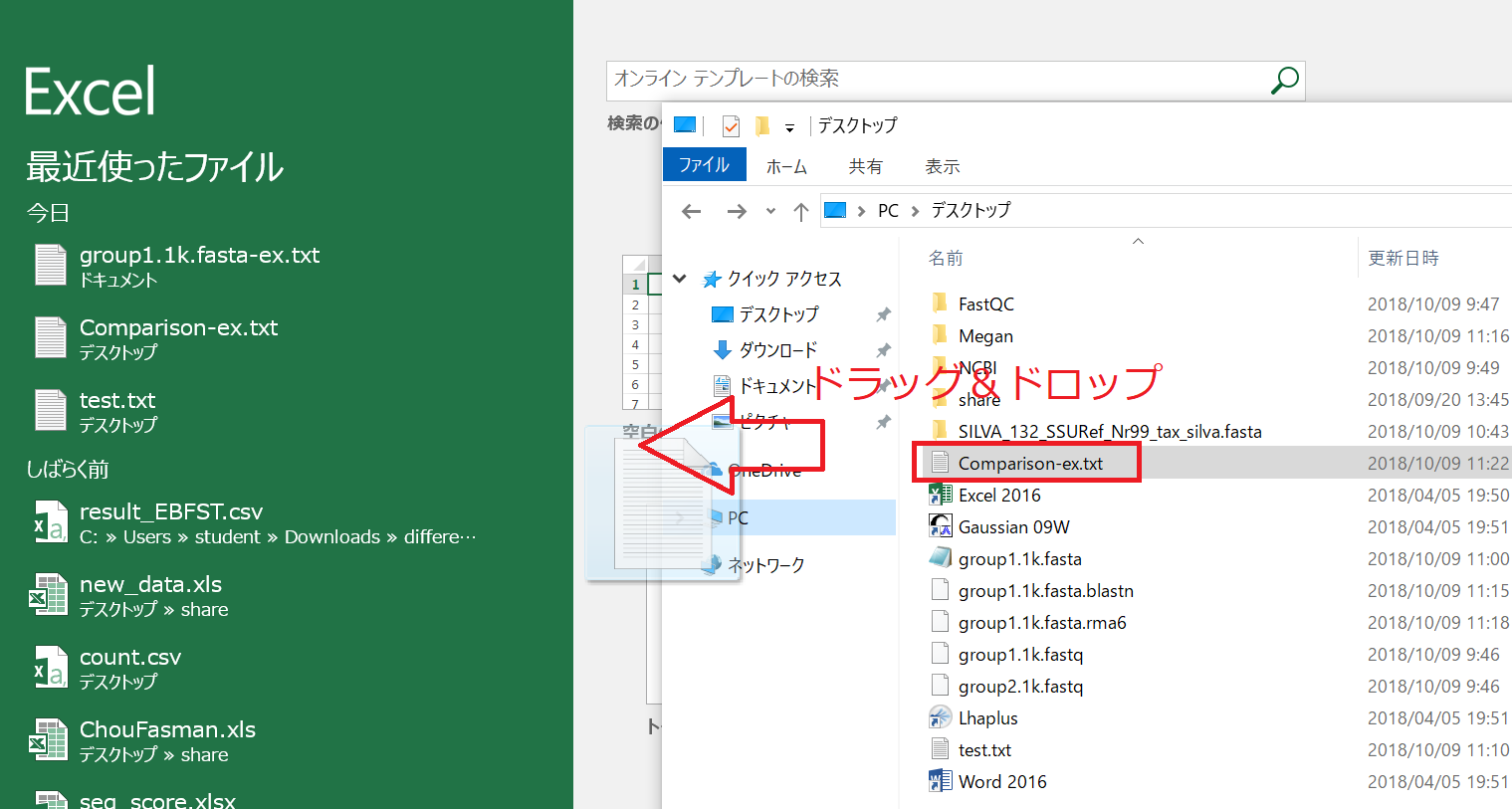
ExportしたファイルをExcelで開くには、WindowsではExcelを起動しておき、ExportしたファイルをExcel上にドラッグアンドドロップすれば良い。
Macの場合は、直接ファイルを開くと文字化けするので、いったんテキストエディットなどで開いてから、中身のテキストをExcelにコピー&ペーストすると良い。
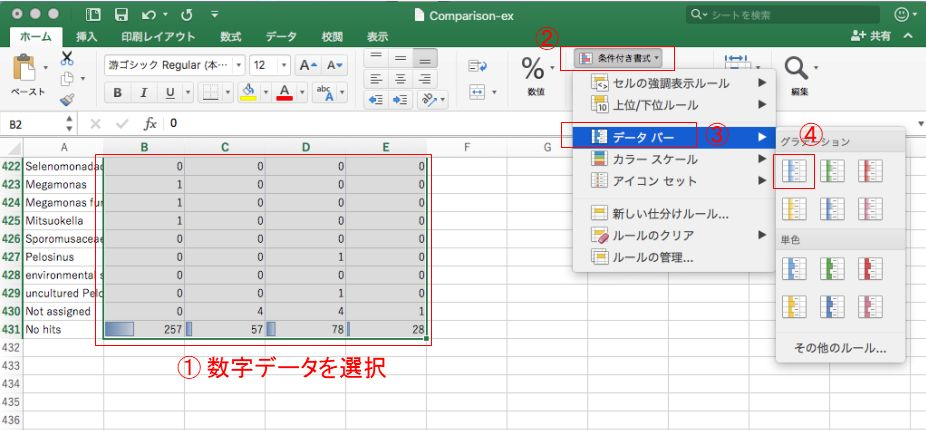
Excelでデータの概要を把握するのに役立つテクニックとして、条件付き書式を設定することで、データの大小を一目でわかるようにできたりします。
そのほか、「データ」→「フィルター」を使ってみたり、グラフを描いてみたり、Excelの機能で各サンプルでリード数の多い順番に生物種名を並び替えてみるなど。
来週の内容
各班次の内容について「目的」、「方法」、「結果」、「考察」の4つのパートを明確に区別してプレゼンテーションを作成してください。班ごとに発表し、発表時間は質疑応答を入れて30分。
X班.制限酵素処理 X班.食品の品種判別 by サンガー X班.三四郎池のeDNA X班.三四郎池, 発酵食品のメタゲノム
課題
来週のプレゼン資料の完成版をファイルに保存して、発表時に提出すること。