rRNA配列を用いた魚種判別、系統樹解析
サンガーシーケンスのデータを開いて、データベースで相同性検索を行い、肉片の魚種を推定する。さらにシーケンスした配列を含めた魚類の系統樹を作成する。
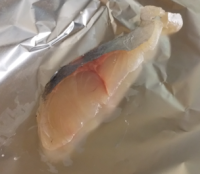
使用した切り身の写真
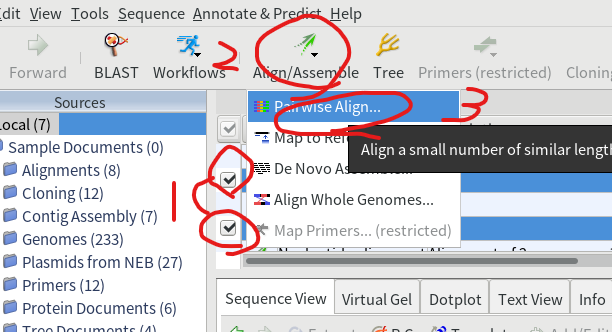
A. サンガーシーケンス配列解析、および系統樹作成のプログラムであるGeneiousの体験版をインストール
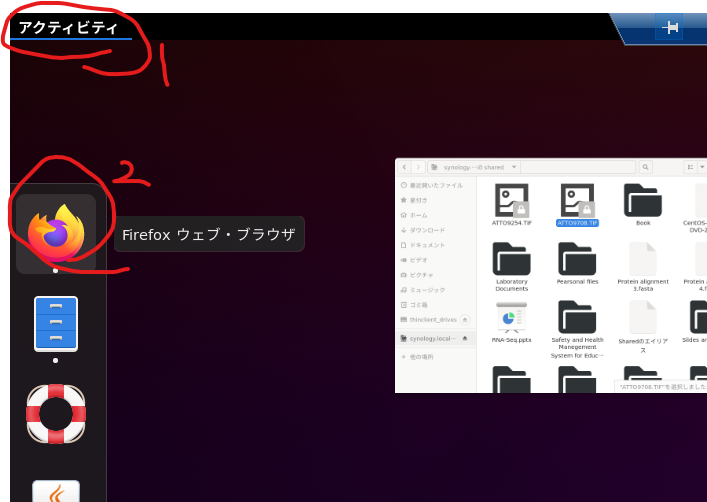
1.ウェブブラウザ(Firefox)を開く。
2.とりあえず、今見ているこのページをFirefoxで開いておきましょう。皆さんのPCとサーバ間では文字のコピー&ペーストが行えるはずです。
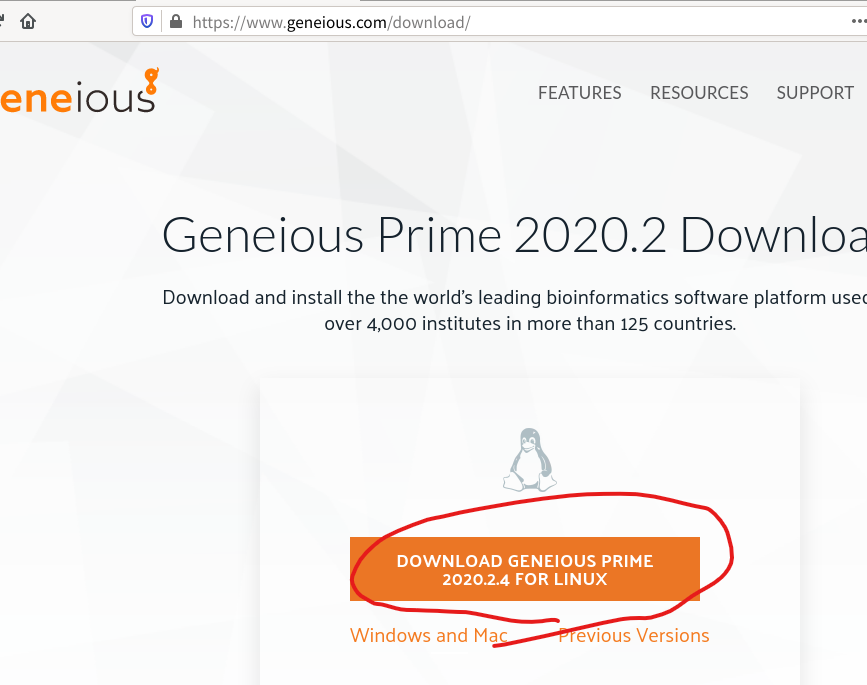
3.Geneiousのインストールファイルをダウンロードする。下記のページの「Download…」ボタンをクリックします。(スクリーンショットは一部古くなっている箇所もあり、バージョンがずれていることがあります。)
https://www.geneious.com/download/
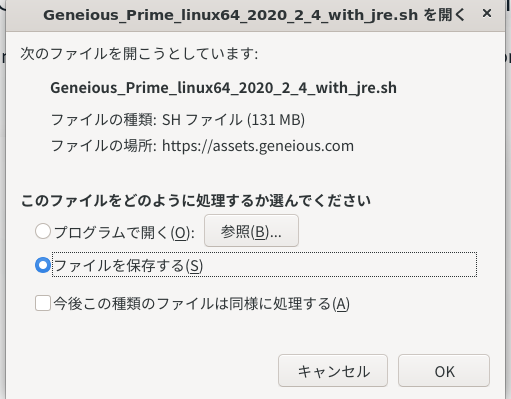
4.「ファイルを保存する」で「OK」を押します。(WindowsやMacの人は、それぞれ適当にファイルを保存したら、ダブルクリックするなりしてインストールを進めてGeneiousを起動し、9.まで進んでください。)
5.ターミナルを開くために、「アクティビティ」→ 「…」→「端末」アイコンをクリックしてターミナルを開きます。
6.ダウンロードしたGeneiousのインストーラを実行するために下記のコマンドを入力します。
#ダウンロードフォルダに移動する cd ダウンロード
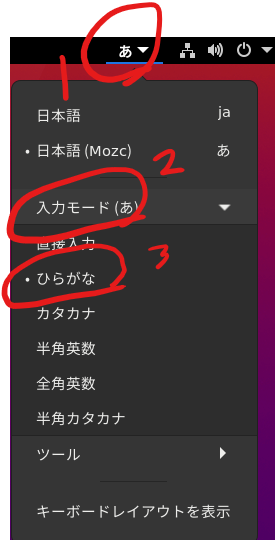
この時、日本語入力と切り替えられないときは画面右上のボタンをクリックして切り替えます。
#ダウンロードしたシェルスクリプトを実行する sh Geneious_Prime_linux64_with_jre.sh
ターミナルで良く使用するキー、コマンドとしては
・カーソルキーの↑↓でコマンドの履歴を表示する ・Tabキーを押して途中まで入力したコマンド、ファイル名などを補完する ・ls ・・・ 現在のフォルダの中にあるファイル一覧を表示 ・cd ・・・ ディレクトリを移動する ・more / less ・・・ ファイルの中身を表示する
7.Geneiousのインストーラに従ってインストールを行います。
8.そのままGeneiousを起動します。
9.「Trial Later」をクリックします。
B. データベースで相同性検索を行う
1.下記のzipデータをダウンロードし、アーカイブマネージャーで開いて、「展開」をクリックし、適当な場所にファイルを解凍する。とりあえず使うのは各自担当したサンプルのフォワードとリバースのシーケンスデータ。
http://suikou.fs.a.u-tokyo.ac.jp/yosh_data/2021jissyu/2021sanger.zip
2.Geneiousでシーケンスファイルを開く。「Sources」をLocalなど適当な場所を選択しておいて、「File」→「Import」→「From File…」をクリックし、先ほど解凍したシーケンスファイルを選択して、「Import」をクリックする。
3.拡大すると、サンガーシーケンスの波形を見ることが出来る。
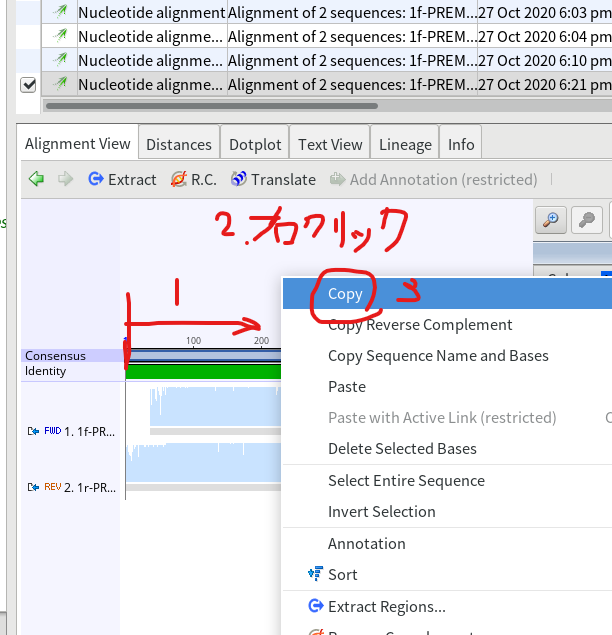
4.綺麗に読めた部分だけ抜き出して、別ファイルに保存する。まずは綺麗に読めた部分をドラッグ&ドロップで選択し、右クリックしてコピーを選択する。
5.リストの適当なところで右クリックして、Pasteを押す。
6.フォワードとリバースで抜きだした配列を選択し、アライメントを実行する。
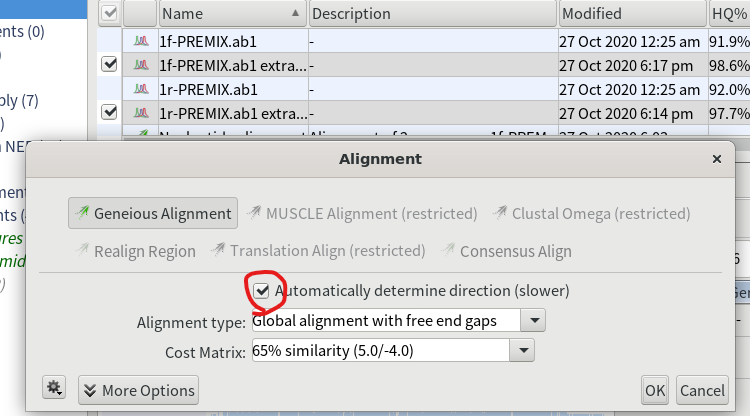
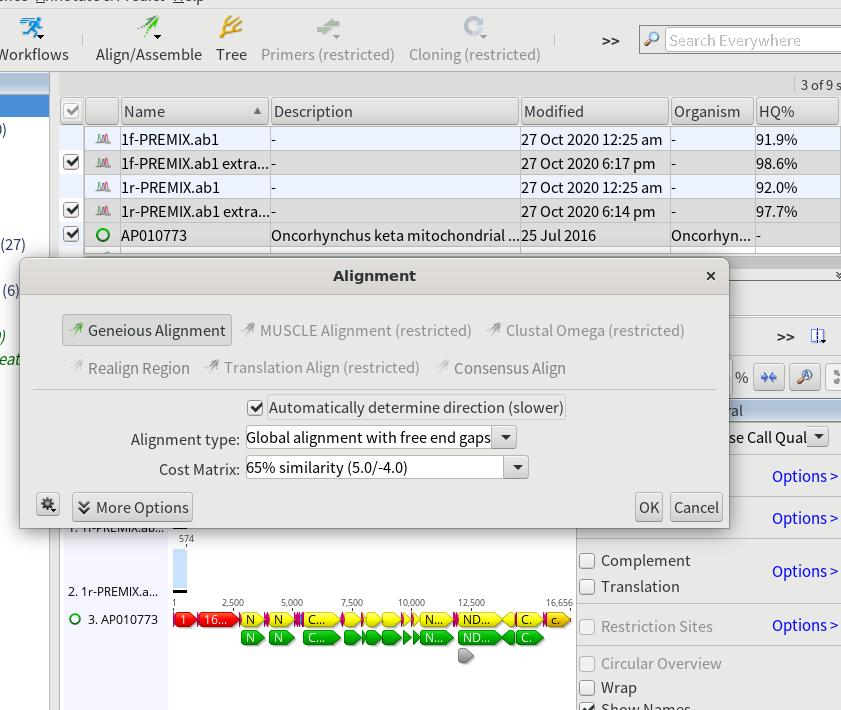
7.自動で配列の向きを合わせてくれるように「Automatically determine direction (slower)」にチェックを入れておく。
8.Consensus配列をドラッグ&ドロップで選択し、右クリックしてCopyする。
9.下記のNCBI BLASTのページを開いて、「nucleotide → nucleotide」(通称blastn)を開いて、コピーした配列をクエリーに張り付ける。Databaseにnr/ntが選択されていることを確認して、BLASTをクリックする。nr/ntはGenbankに登録された配列の冗長さを除去した(non-redundantな)データベースで、幅広い生物種・遺伝子が網羅的にバランスよく登録されていて、まずはここで配列を検索する研究者が多い。
https://blast.ncbi.nlm.nih.gov/Blast.cgi
10.そのほか、魚類のミトコンドリア専用のデータベースとして、東大岩崎研でメンテナンスされているMitoFishデータベースがある。
http://mitofish.aori.u-tokyo.ac.jp/
こちらは2020年10月現在、2,939種の魚の完全長のミトコンドリアが登録されている。NCBI NTデータベースと、MitoFishデータベース両方で検索して違いを比較してみる。
11.ミトコンドリアの配列をデータベースからダウンロードし、シーケンスデータとアライメントを行い、シーケンスした領域がどこだったのか確認する。
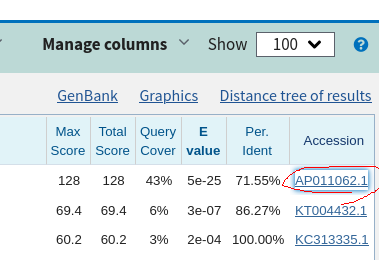
NCBI、MitoFishどちらも結果を適当にクリックしていると、ヒットした配列のAccession番号のリンクを見つけることが出来るはず。Accession番号を開くと、GenBankに登録されている配列をアノテーション付きで表示することが可能である。
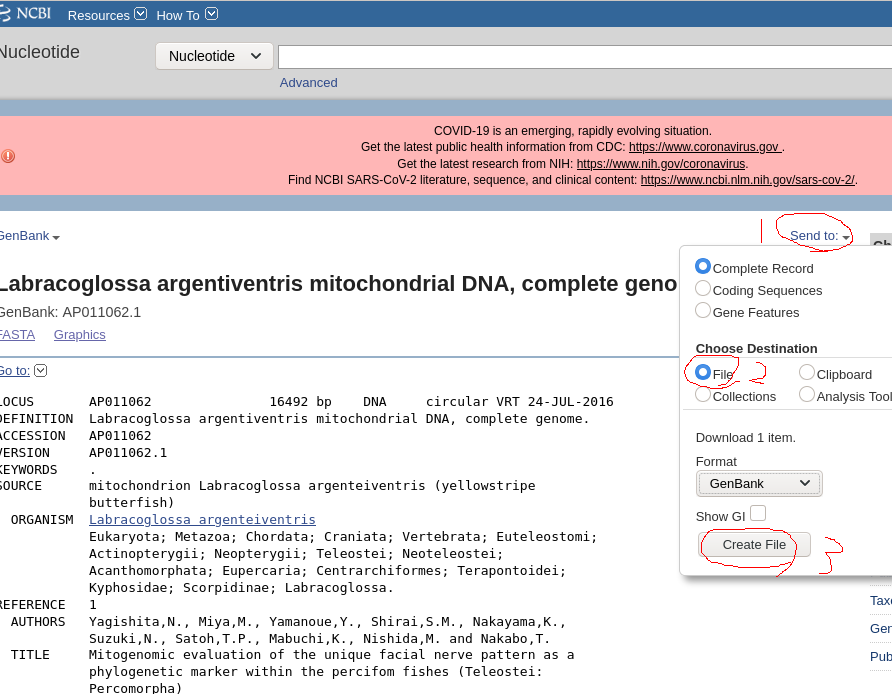
12.シーケンスデータと相同性のある完全長ミトコンドリアの配列をGenBankで開き、GenBank形式でファイルに保存するを選んでダウンロードする。
13.Geneiousで「File」→「Import」→「From File…」をクリックし、先ほどダウンロードしたGenBankファイルを開く。
14.シーケンスデータと、インポートしたGenBankファイルを選択し、「Align/Assemble」→「Multiple Align…」をクリックする。「Automatically determine direction (slower)」にチェックを入れて「OK」を押す。
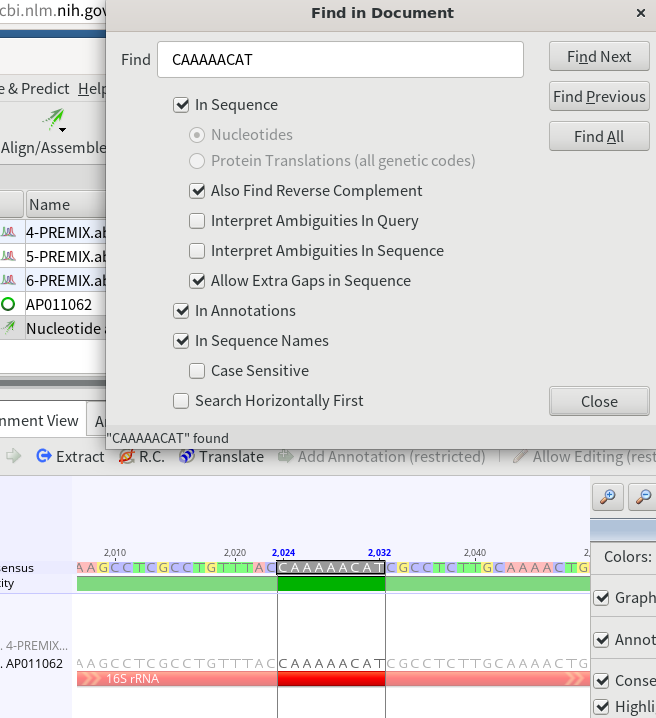
15.Alignment ViewでCtrl+Fを押すと、配列を検索する画面が開くので、下記のプライマーの配列を張り付けて検索してみる。もしヒットしない場合は、プライマーの配列が保存されていない可能性もあるため、適当に検索する配列を短くしてヒットするかどうかを見ていく。ヒットする配列は基本的には16S rRNAの中にあるはずである。
16SarL CGCCTGTTTATCAAAAACAT 16SbrH CCGGTCTGAACTCAGATCACGT
16.プライマーの配列から何塩基くらい離れたところからシーケンスが開始されているか、シーケンスデータとリファレンス配列が不一致な場所があれば、波形が綺麗に読まれているかどうかを確認する。
自分の担当したサンプル番号の魚を上の切り身の写真と、配列の検索結果から推定し、Zoomのチャットに書き込む。
C. 系統樹作成
ミトコンドリア16S rRNAによる魚類の系統樹を作成し、魚類の中での系統関係を解析する。
1.下記のファイルには46種の生物のミトコンドリア16S rRNA配列が含まれているのでダウンロードする。
http://suikou.fs.a.u-tokyo.ac.jp/yosh_data/2020jissyu/16S-46sp.fasta.zip
2.Geneiousの「File」→「Import」→「From File…」からダウンロードしたファイルを開く。その際、「Nucleotide sequences」→「Keep sequences separate」を選んでおく。
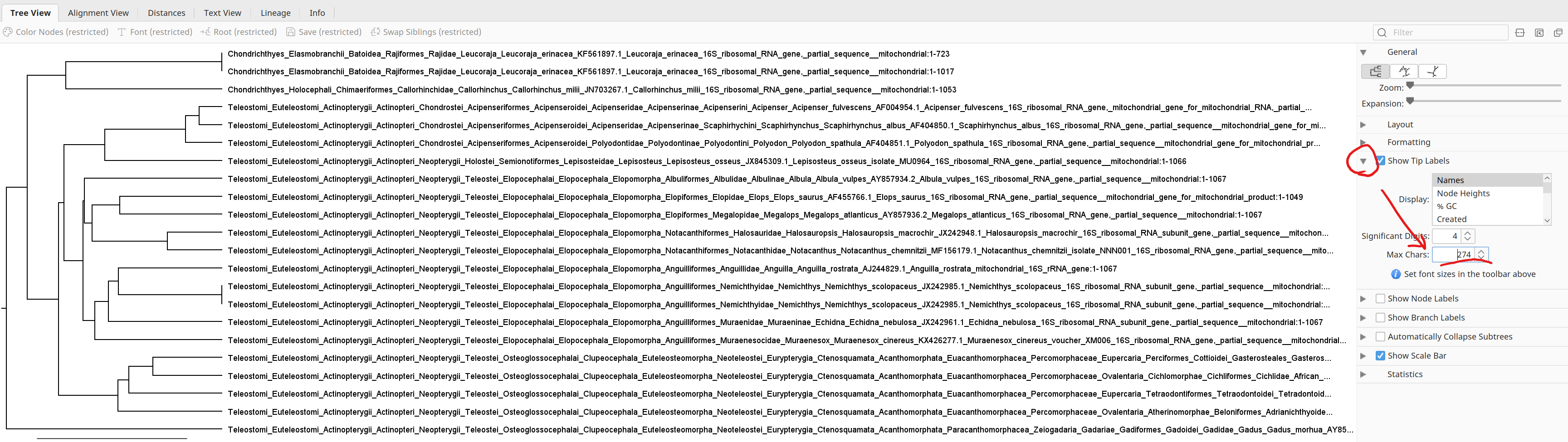
3.各自担当した魚肉の配列と、追加した46種の配列を全て選択して、「Align/Assemble」→「Multiple Align…」をクリックし、マルチプルアライメントを作成する。(しばらく時間がかかる。)
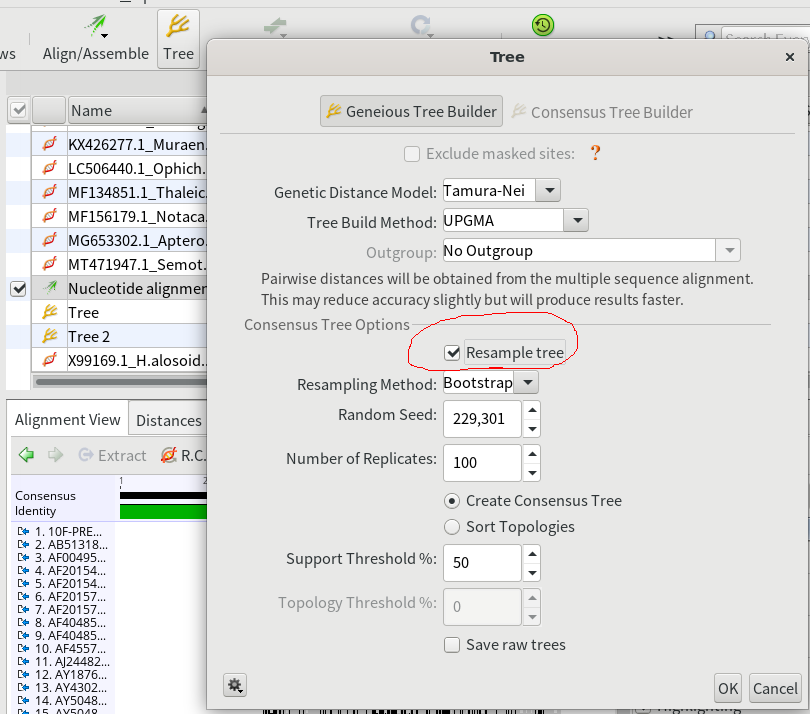
4.マルチプルアライメント結果を選択した状態で、「Tree」をクリックし、UPGMAによる系統樹を作成する。その際各分岐の確からしさをブートストラップ法によって求めるため、「Resample tree」にチェックを入れて「OK」を押す。
5.作成された系統樹には、大分類群から分類名が書かれているため、そのままでは見えづらいので、Tip Labelsを開いて、Max Charsを30→200などに変更して分類名を表示してみる。そうすると、「Chondrichthyes(軟骨魚綱)」と硬骨魚類の属する「Teleostomi(真口亜綱)」で分かれているのが見られるはず。
6.(余裕があれば)Genbankに登録された配列の中から、興味のある魚種のミトコンドリア16Sを抽出してGeneiousに取り込み、系統樹を作成する。
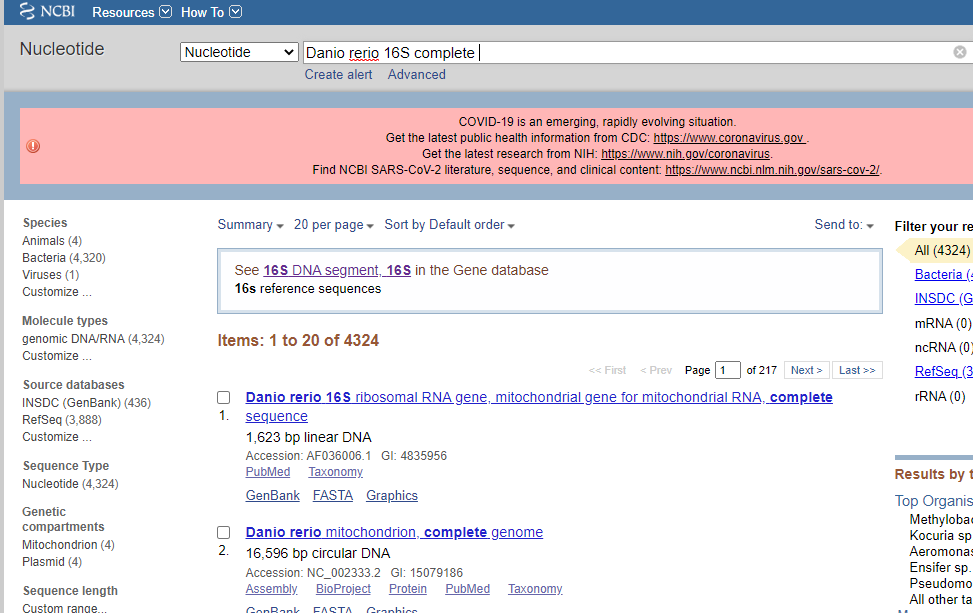
https://www.ncbi.nlm.nih.gov/nucleotide/
例えばゼブラフィッシュの16Sを探したい場合は、「Danio rerio 16S complete」などで検索してみる。
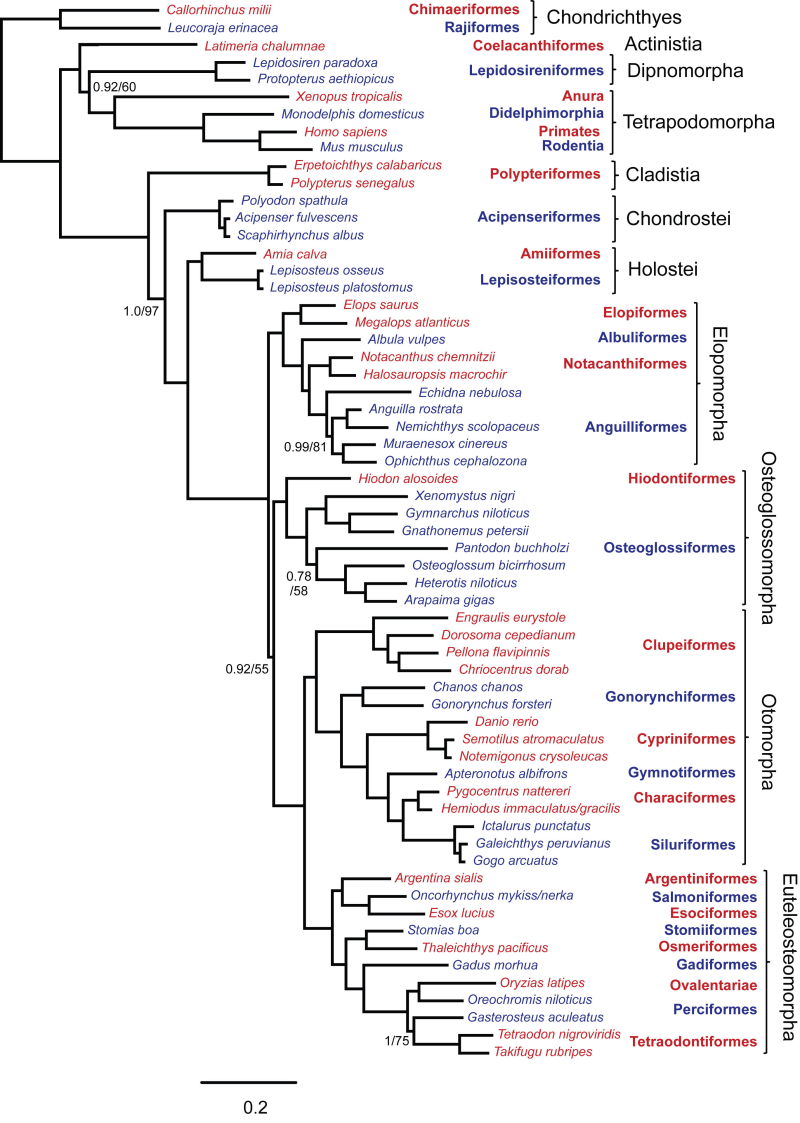
作成した系統樹は既知の系統樹と一致するか、一致しない場合は理由を考えてみる。
既知の系統樹の例:
D. 検討項目の例
- プライマーの配列から何塩基くらい離れたところからシーケンスが開始されているか。
- NCBI NTデータベースとMitoFishデータベースの相同性検索結果に違いはあったか。どちらが望ましい結果だったか。
- 作成した系統樹は既存の分類群(科・属・種名など)がどの程度正しく描けていたか。