2019年 水圏生物科学実験III
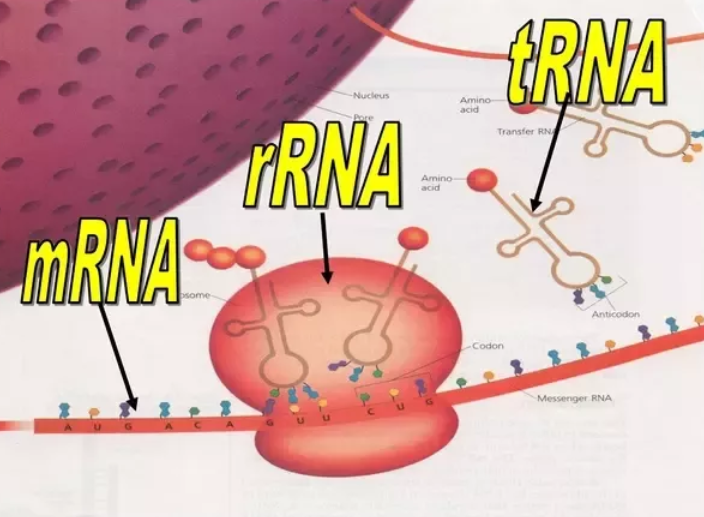
リボソームについて
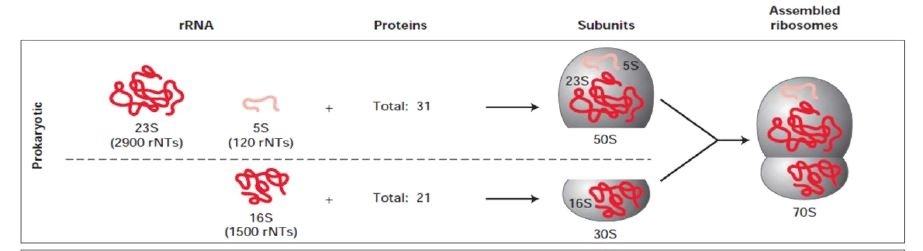
リボソームはRNAとタンパク質の複合体であり、原核生物の場合、大きく50Sと30Sのサブユニットに分かれる。30S複合体の中には16S rRNAが含まれる。
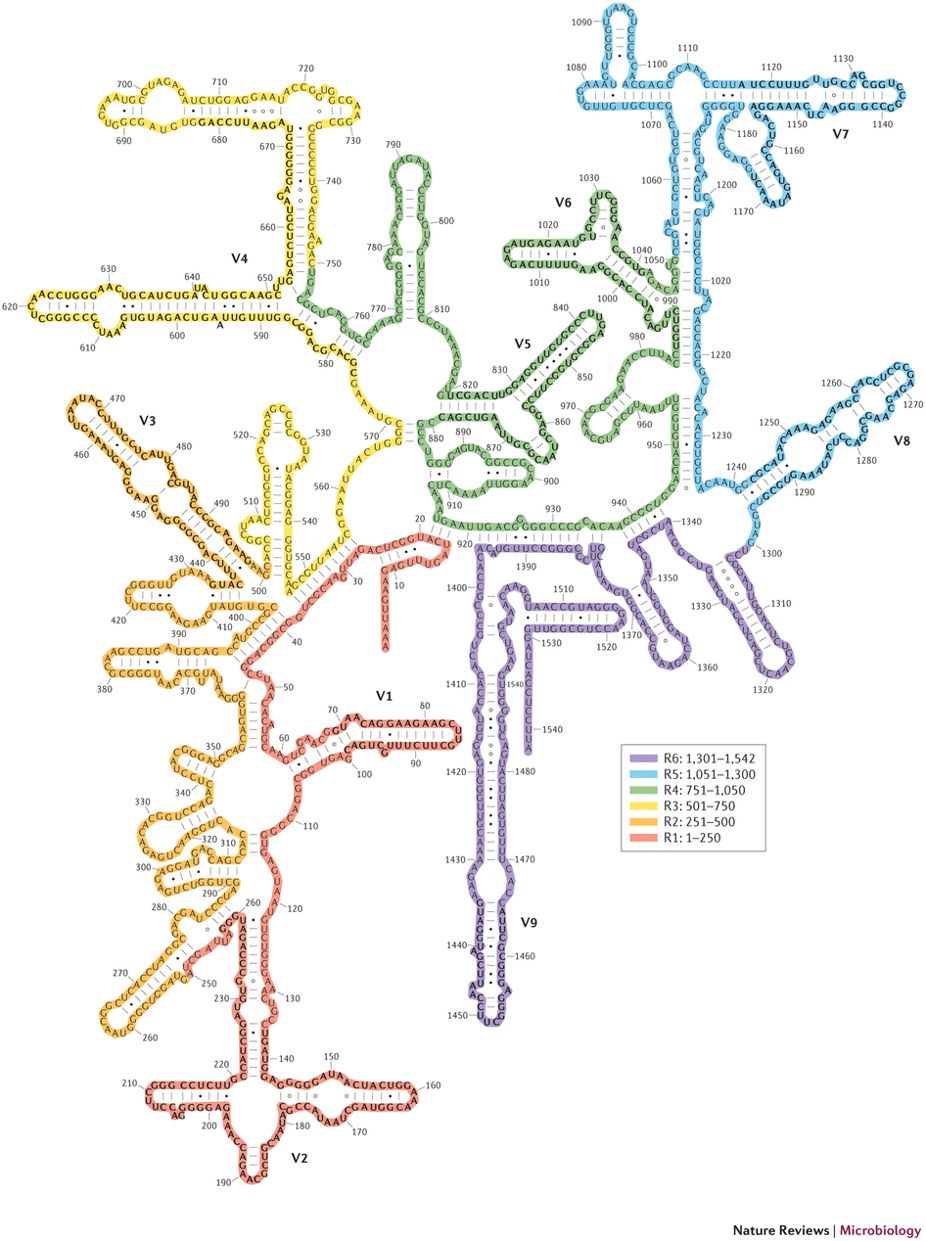
16S rRNA
16S rRNAは保存性の高いConserved Regionsと、変化の多いVariable Regionsが交互に出現する。
rRNAはmRNAとは異なり、二次構造を取ることが知られている。
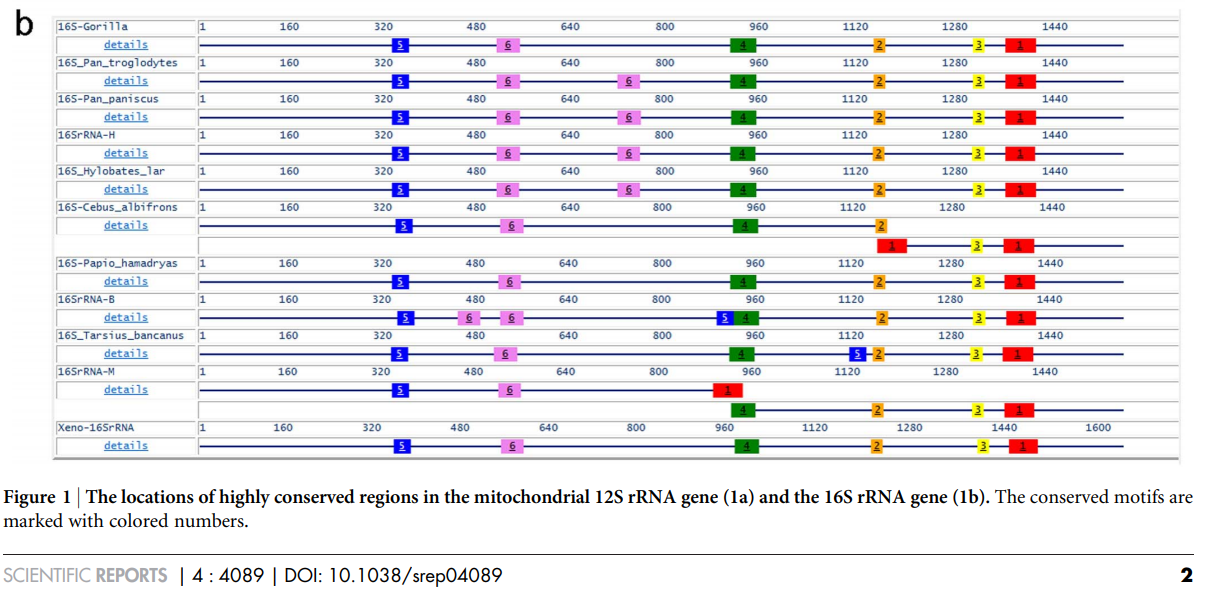
12S rRNAと16S rRNA
12S rRNAは真核生物のミトコンドリアにコードされている遺伝子で、16S rRNAに相同な遺伝子である。
学生実習で使用したプライマー
16S, 12Sの保存性の高い領域に設計したプライマーを使用している。具体的には次の配列を使用している。
| 名前 | 配列 | 生物種 | 増える長さ |
| ミトコンドリア16S一部 Forward (16SAR-L) | CGCCTGTTATCAAAAACAT | 脊椎動物,節足動物,軟体動物等 | 600 bp程度 |
| ミトコンドリア16S一部 Reverse (16SBR-H) | CCGGTCTGAACTCAGATCACGT | 脊椎動物,節足動物,軟体動物等 | 600 bp程度 |
| バクテリア16S全長 Forward (27F) | AGAGTTTGATC(A/C)TGGCTCAG | バクテリア全般 | 1.5 kbp程度 |
| バクテリア16S全長 Reverse (1492R) | GGTTACCTTGTTACGACTT | バクテリア全般 | 1.5 kbp程度 |
| ミトコンドリア12S全長 Forward | AHTTAYACATGCAAGTHTCM | 魚類 | 1 kbp弱 |
| ミトコンドリア12S全長 Reverse | YRCACYTTCCRGTACRCTTA | 魚類 | 1 kbp弱 |
| ミトコンドリア12S MiFish Forward (MiFish-U-F) | GTCGGTAAAACTCGTGCCAGC | 魚類 | 200 bp程度 |
| ミトコンドリア12S MiFish Reverse (MiFish-U-R) | CATAGTGGGGTATCTAATCCCAGTTTG | 魚類 | 200 bp程度 |
配列の相同性検索について
相同性検索について
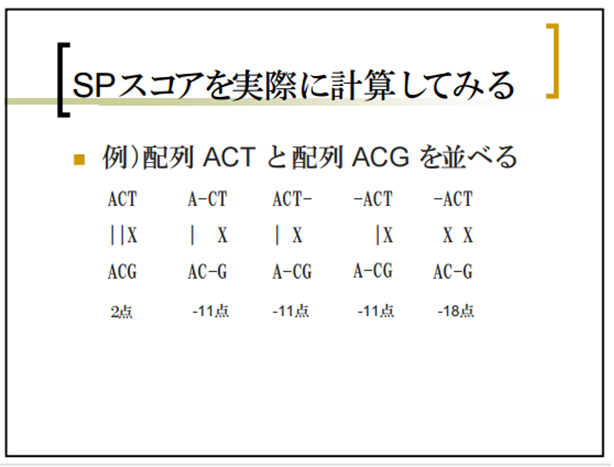
最も似ているアライメントを効率よく求めるSmith-Watermanアルゴリズム
相同性検索を高速に行うBLASTプログラム
BLASTは、相同性検索(ホモ ロジーサーチ)を比較的高速に行うプログラムである。厳密な解を提供する Smith-Watermanアルゴリズムを少しヒューリスティックにすることで、完全な厳密解は与えないものの実用的には十分な精度を持ちつつ、 Smith-Watermanよりはるかに高速に検索を実現した。 また、BLASTではペアワイズの相同性検索の結果に対して、偶然そのような配列の一致が起こる期待値e-valueを出力し、閾値以上でデータベースとヒットした結果を出力する。
BLASTの高速化の概要
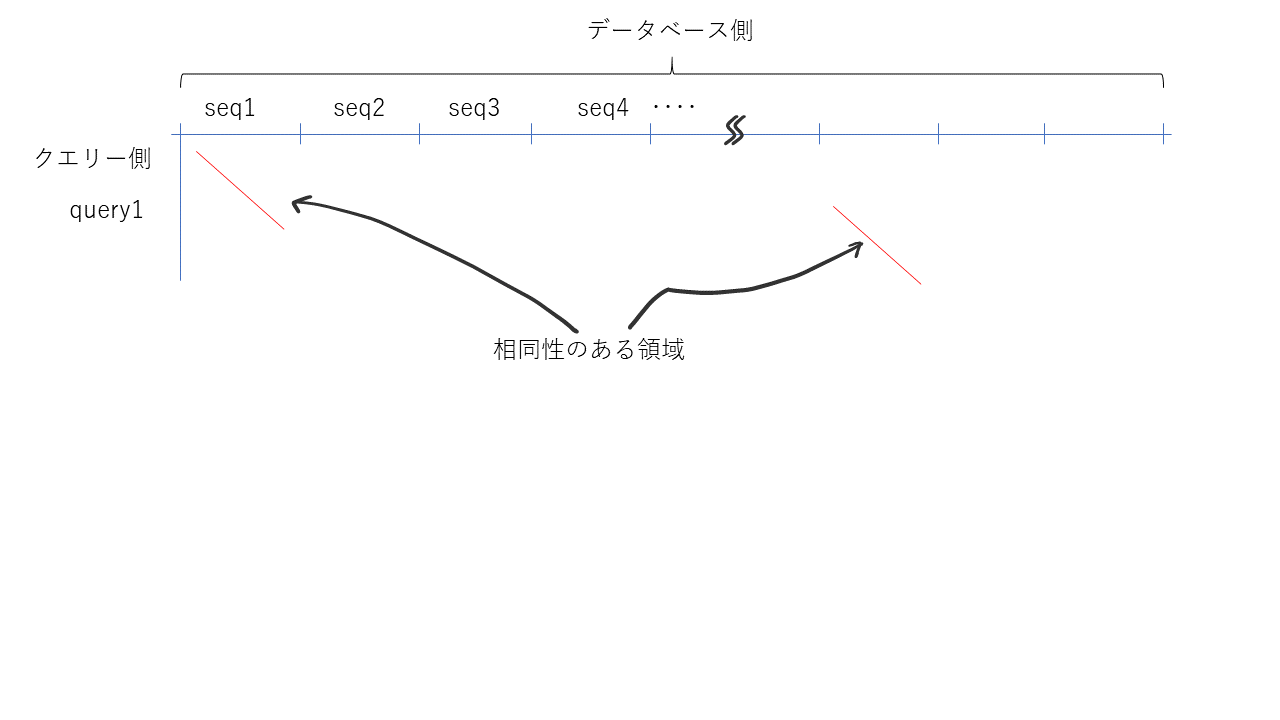
データベース検索する場合、下記のようにデータベース側は100 Gbaseを超える場合もあり、非常に巨大である。しかし、その中で相同な配列というのは通常そんなに多くはない。
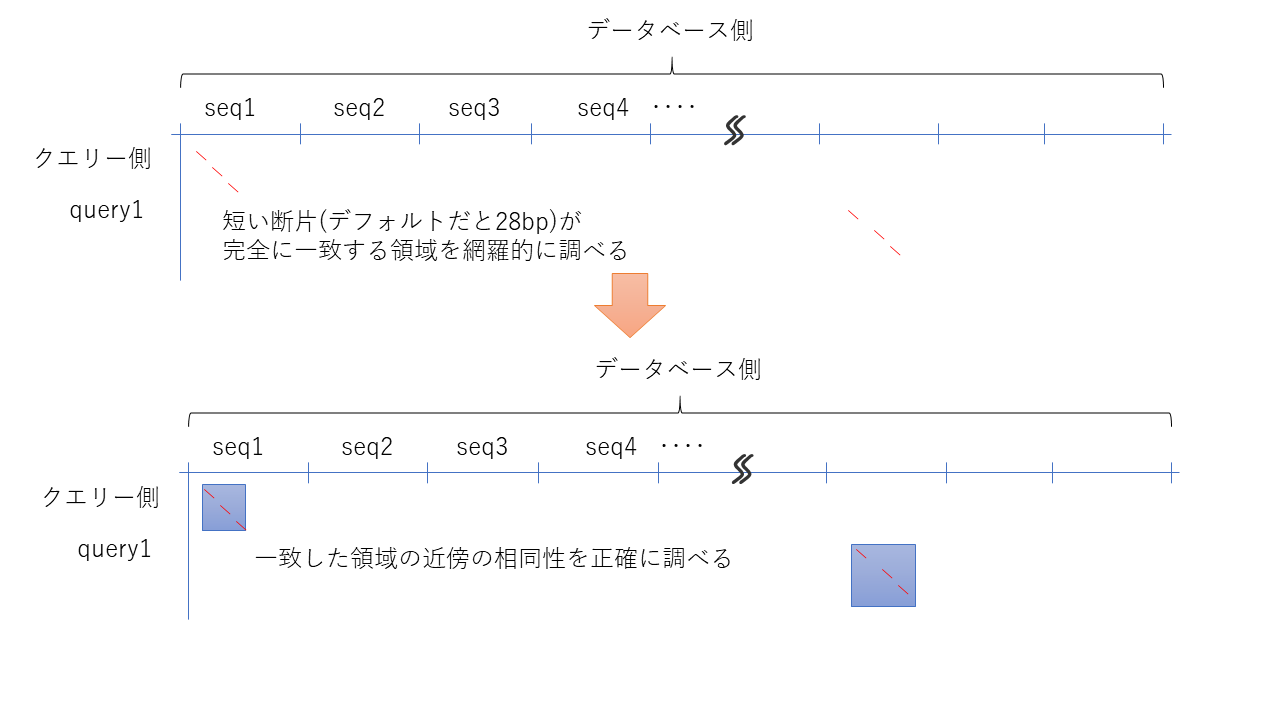
そこで、あらかじめwordサイズで指定した大きさで100%マッチする場所を高速に調べておき、その周辺のみ時間をかけて調べることで高速化している。
このwordサイズはNCBIのWEBサイトで公開されているBLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi )などでは、デフォルトが28 bpと比較的大きめなので、シーケンス精度が悪い場合は注意する必要があるかもしれない。
BLASTプログラムの種類
BLASTでは、問い合わせ配列とデータベース配列の組み合わせから、次の5種類が用意されている。
| プログラム名 | query | db | |
| blastn | DNA | DNA | |
| blastp | protein | protein | |
| blastx | DNA | protein | (DNAはアミノ酸に翻訳して比較) |
| tblastn | protein | DNA | (DNAはアミノ酸に翻訳して比較) |
| tblastx | DNA | DNA | (DNAはアミノ酸に翻訳して比較) |