**文書の過去の版を表示しています。**
2017年 水圏生物科学実験III
本日の概要
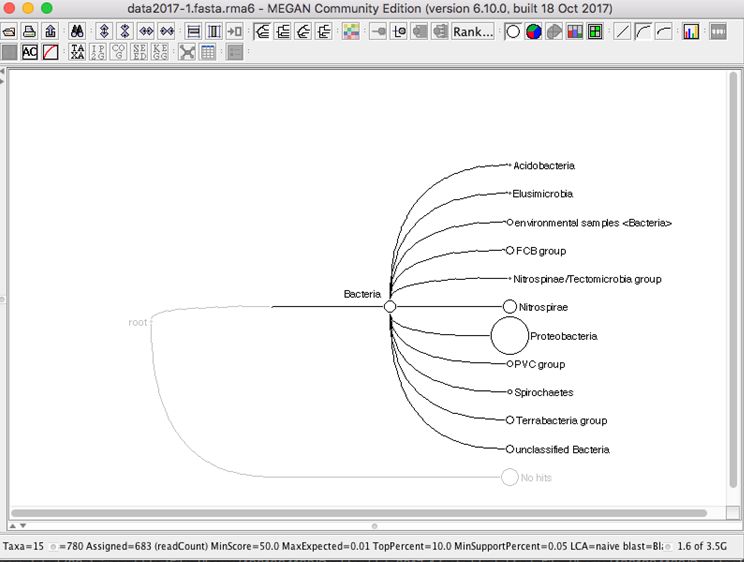
次世代シーケンサーから得られるデータの解析を行う。データはOxford Nanoporeから得られた16S rDNAのアンプリコンメタゲノムデータである。
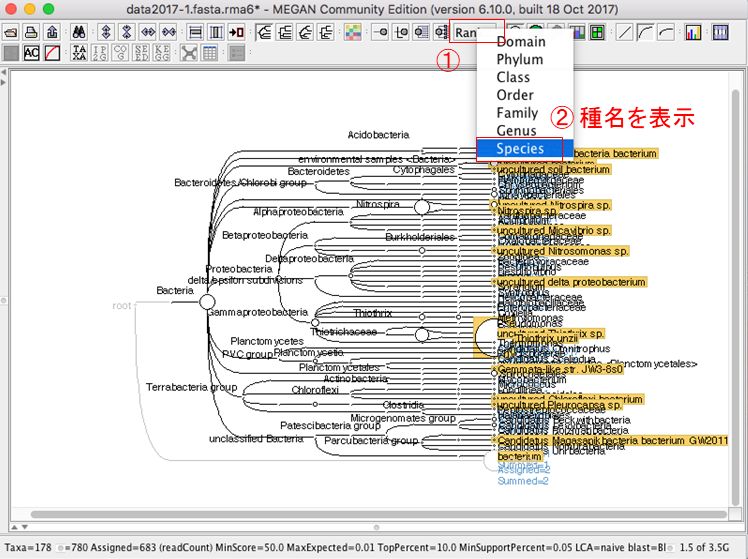
データ解析の概要としては、rDNAのデータベースであるSILVA databaseに対して、Nanoporeのデータの相同性検索を行い、検索した結果をMEGANというツールで統合し、可視化する。
データの説明
16S rDNAについて
準備
ツール・データのダウンロード
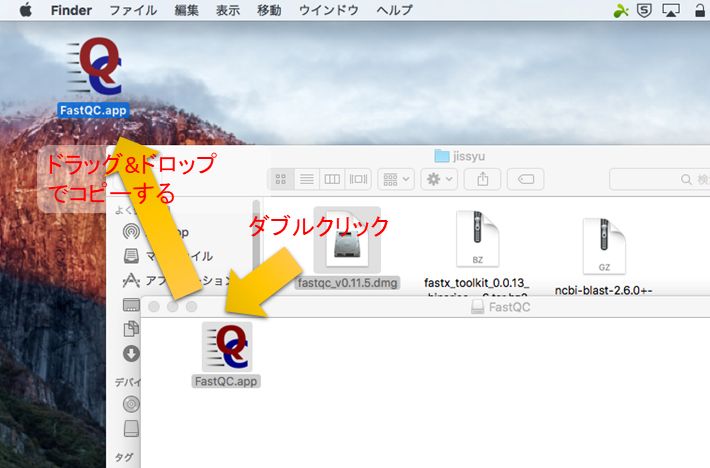
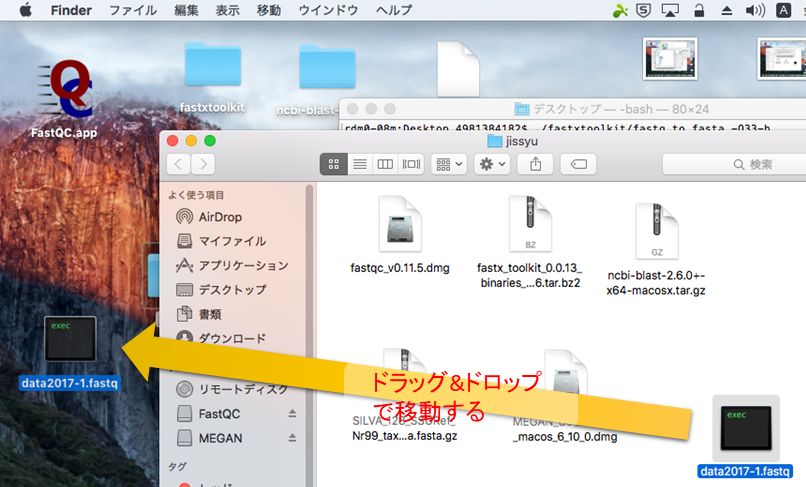
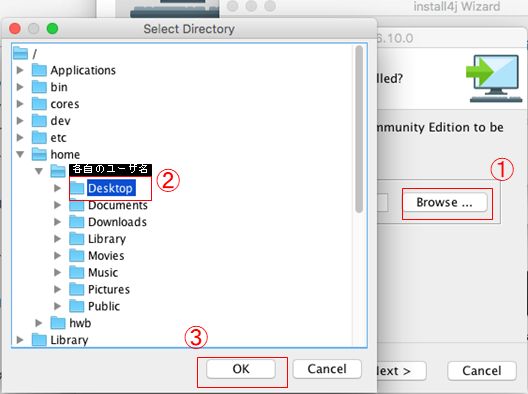
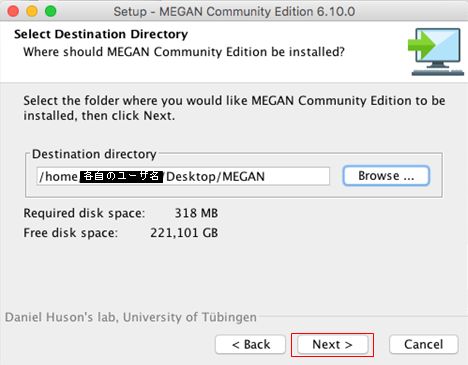
MACでログインしたら、デスクトップに「ダウンロード」フォルダ等の適当なフォルダを作り、その中に下記のファイルをすべてダウンロードする。
- FASTQC ・・・シーケンスデータのクオリティチェックを行う。(https://www.bioinformatics.babraham.ac.uk/projects/fastqc/)
- FASTX-Toolkit ・・・シーケンスデータのトリミングやフィルタリング、変換等を行う。(http://hannonlab.cshl.edu/fastx_toolkit/)
- BLAST ・・・言わずと知れた相同性検索ツール。(https://blast.ncbi.nlm.nih.gov/Blast.cgi)
- SILVA database ・・・リボソームDNA配列を整理したデータベース。(https://www.arb-silva.de/)
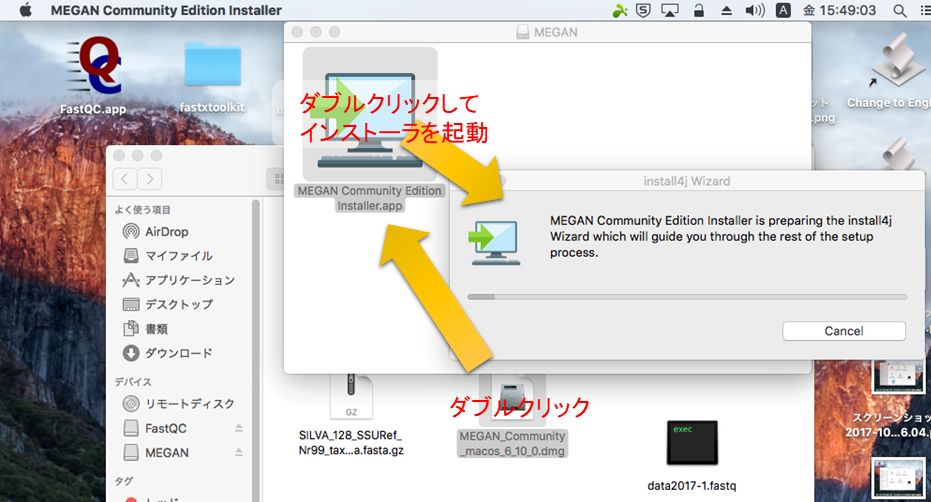
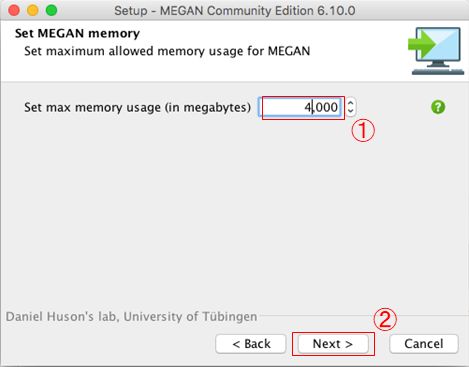
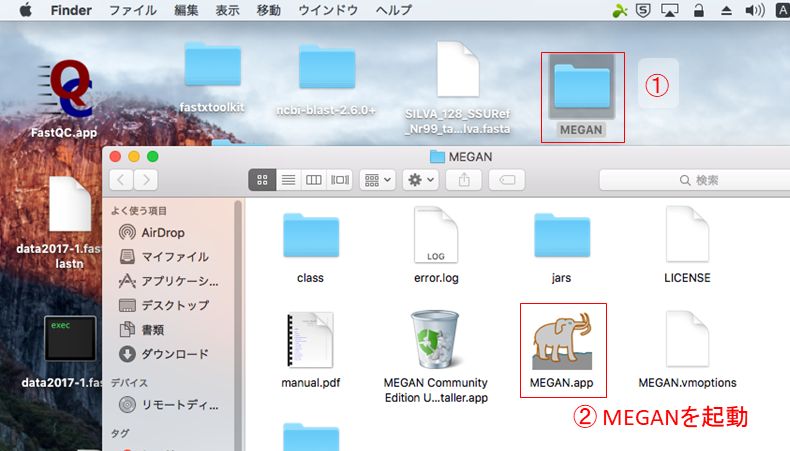
- MEGAN ・・・BLASTの結果から微生物叢情報や機能遺伝子情報へと変換してくれるソフトウェア。KEGGに関しては有償。(http://ab.inf.uni-tuebingen.de/software/megan6/)
シーケンスデータの説明
- FASTQファイルとは
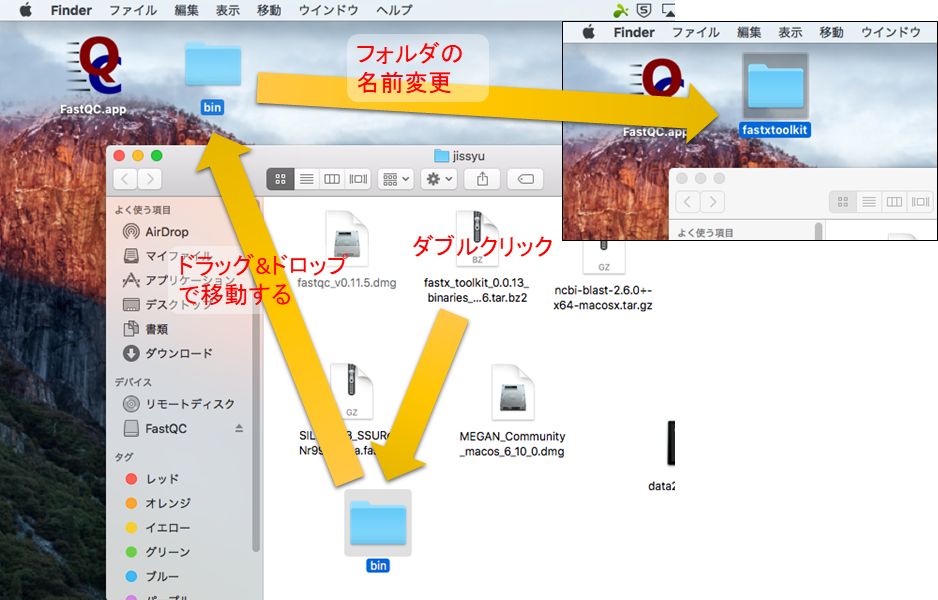
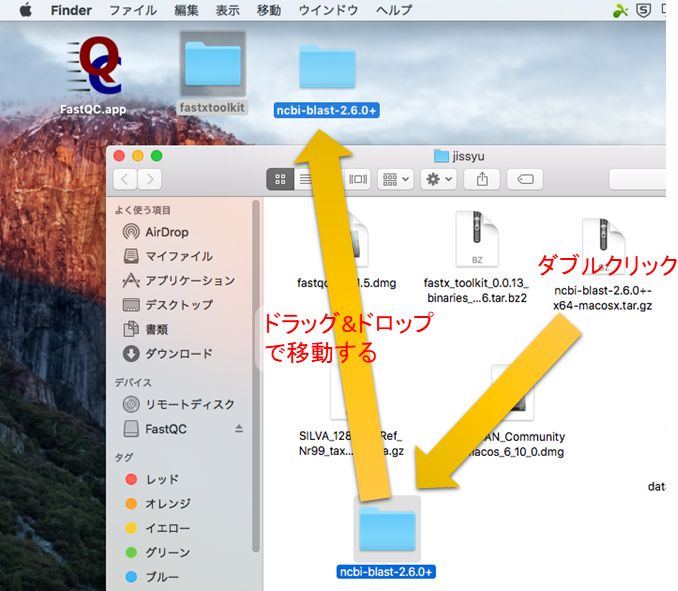
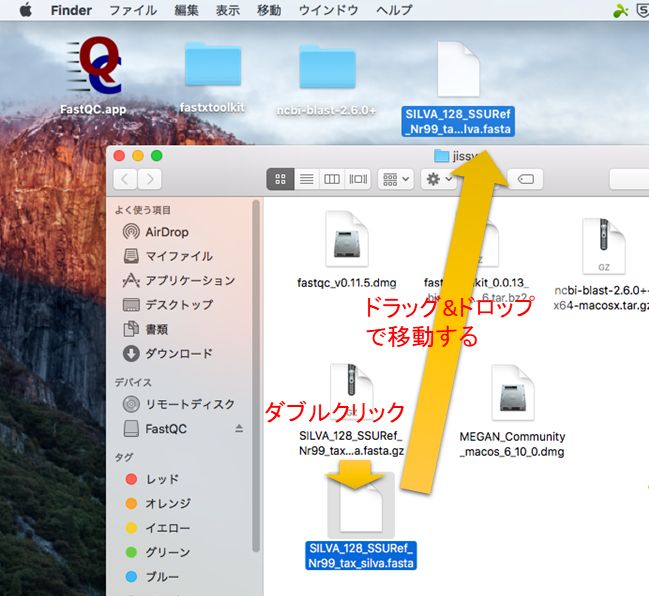
ダウンロードしたツール・データの解凍
データ解析
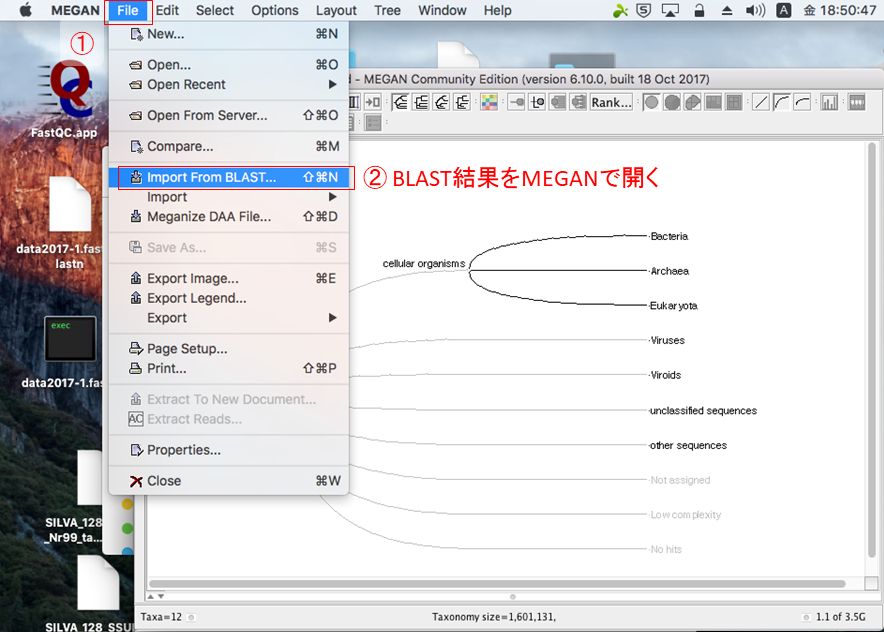
解析手順の概要は、まずFASTQCでシーケンスデータを確認してから、FASTQをFASTAファイルに変換し、SILVAデータベースに対してBLASTを実行する。そして、BLASTの結果ファイルをMEGANで読み込む。
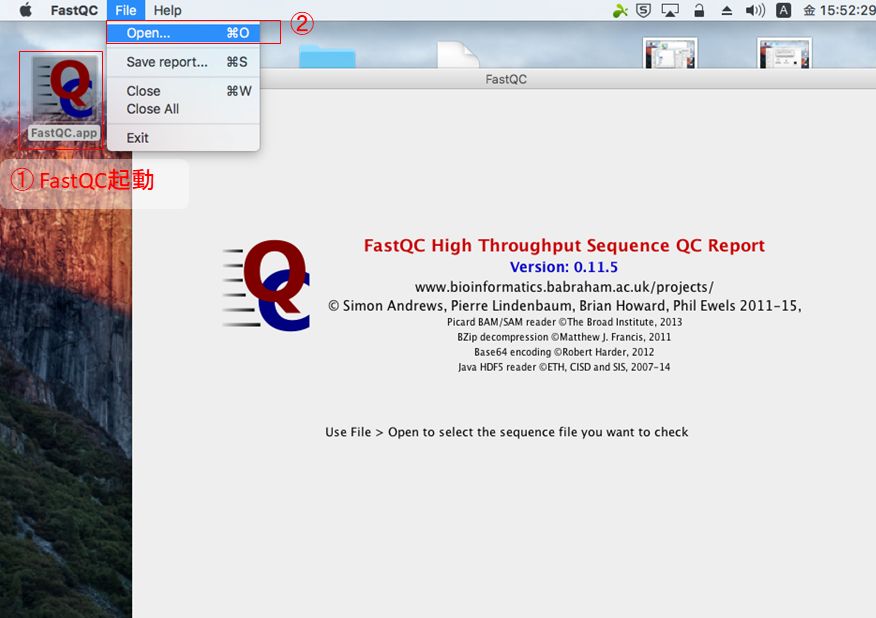
- FASTQC
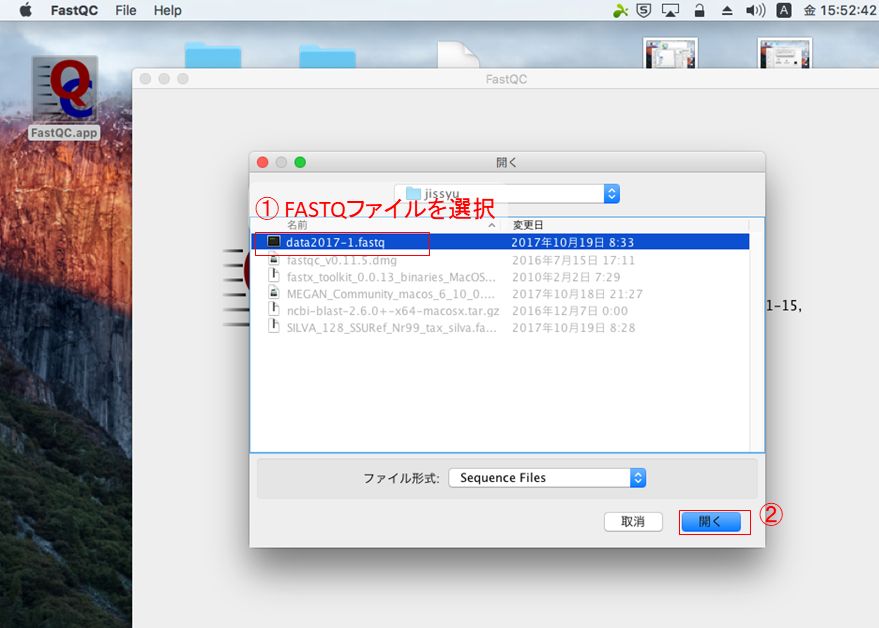
FASTQCを実行し、シーケンスデータを開く。
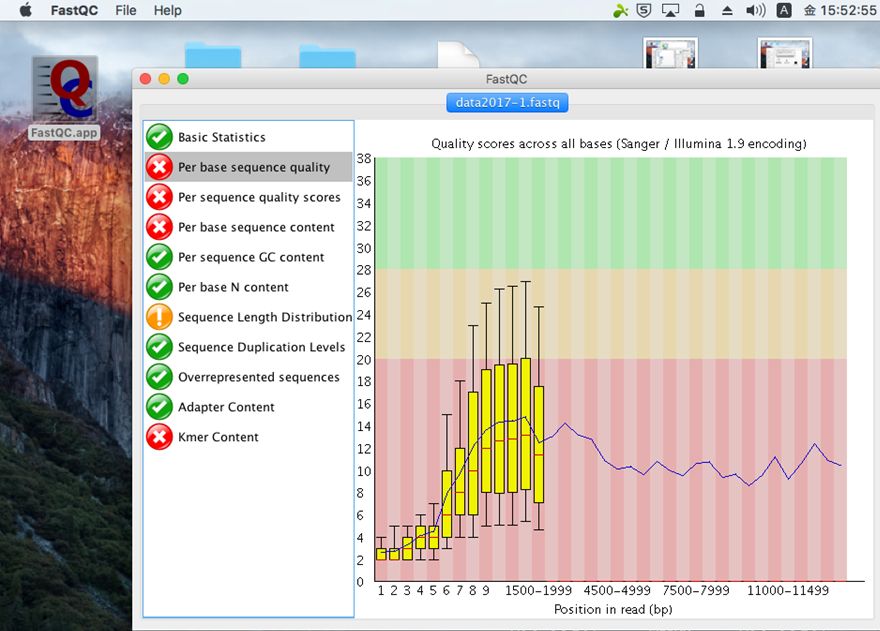
FASTQCは主にIllumina用のクオリティチェックツールであり、Nanoporeのデータに対しては適切な評価ができないので、評価値の〇×は気にしなくてよい。下記はシーケンスデータのクオリティスコアに関する、平均値等の情報。クオリティスコアに関する説明はこちら。Nanoporeのデータだとクオリティスコア10強(精度90%強)となるはずである。
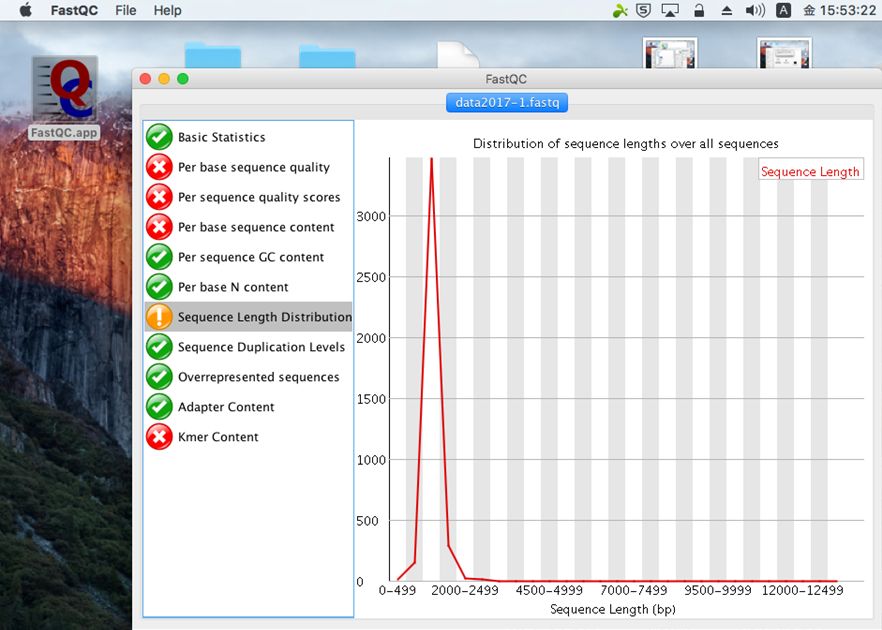
リード長の分布。
- FASTQ→FASTA変換
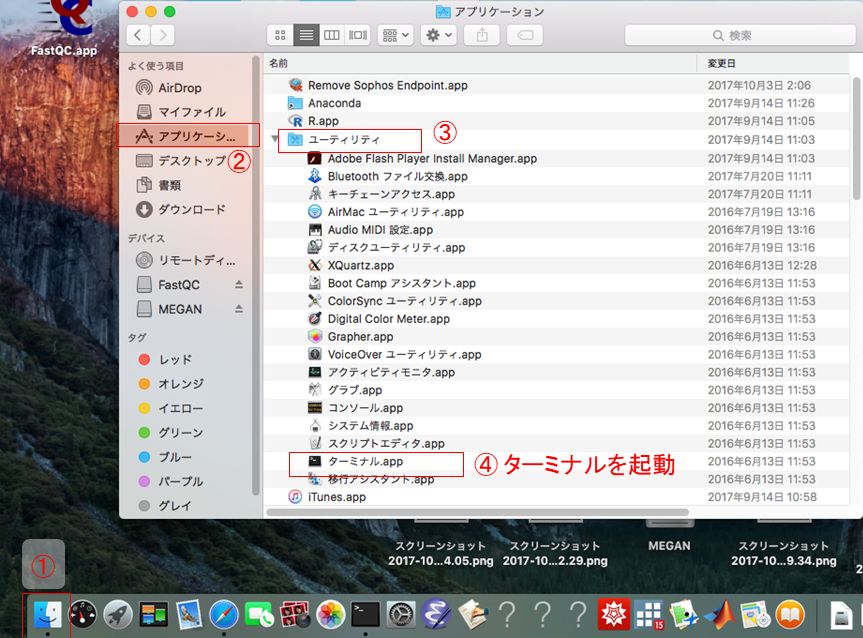
まずはFinderからターミナルを開く。
ターミナルを開いたら、
cd Desktopと入力して、ターミナル内のディレクトリをデスクトップに移す。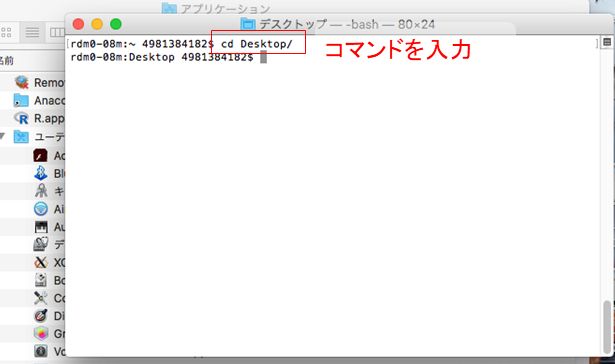
よく使用するコマンドの説明は下記の通り
ls: 現在のディレクトリ内のファイルを一覧表示。cd [ディレクトリ名]: 指定したディレクトリに移動する。less [ファイル名]: 指定したファイルの中身を表示する。表示をやめるときは「q」を押す。ターミナルで「Desktop」に移動したら、下記のコマンドを入力し、FASTQファイルからクオリティを除去したFASTAファイルへと変換する。
./fastxtoolkit/fastq_to_fasta -Q33 -i data2017-1.fastq -o data2017-1.fasta-Q33オプションはクオリティスコアの種類を示すオプションで、おまじないと思ってとりあえず書いておくこと。
-i は入力のFASTQファイル、-o は出力のFASTAファイルの名前を書く。
- BLASTデータベース作成
次にSILVAのrDNAの配列がすべて含まれるFASTAファイルから、BLASTのデータベースを作成する。
./ncbi-blast-2.6.0+/bin/makeblastdb -in SILVA_128_SSURef_Nr99_tax_silva.fasta -dbtype nucl-in には入力となるFASTAファイルを指定する。
-dbtype はFASTAファイルがDNA配列であれば「nucl」、アミノ酸配列であれば「prot」を指定する。
- BLAST検索
FASTA形式に変換したシーケンスデータをクエリーとして、SILVAデータベースに塩基配列の相同性検索を行う。
./ncbi-blast-2.6.0+/bin/blastn -db SILVA_128_SSURef_Nr99_tax_silva.fasta -query data2017-1.fasta -num_threads 4 > data2017-1.fasta.blastn-db には、BLASTデータベースファイルを作成したFASTAファイルを指定する。
-query にはデータベースに対して相同性検索を行いたい配列が含まれるFASTAファイルを指定する。ここではFASTAファイルに変換したシーケンスデータを指定する。
-num_threads には並列計算時に使用するCPUの数を指定する。ECCSでは1台当たり4コアあるので、4を指定。
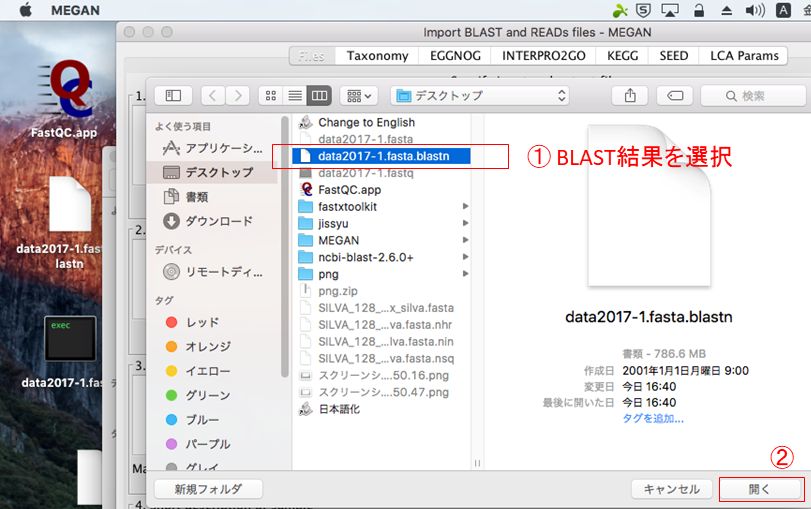
> の後ろには出力ファイルの名前を書く。(拡張子はMEGANに読み込ませるために「.blastn」とすること)